Матричная РНК


Ма́тричная рибонуклеи́новая кислота́ (мРНК, синоним — информацио́нная РНК, иРНК) — РНК, содержащая информацию о первичной структуре (аминокислотной последовательности) белков[1]. мРНК синтезируется на основе ДНК в ходе транскрипции, после чего, в свою очередь, используется в ходе трансляции как матрица для синтеза белков. Тем самым мРНК играет важную роль в «проявлении» (экспрессии) генов.
Длина типичной зрелой мРНК составляет от нескольких сотен до нескольких тысяч нуклеотидов. Самые длинные мРНК отмечены у (+)оц РНК-содержащих вирусов, например пикорнавирусов — однако следует помнить, что у этих вирусов мРНК образует весь их геном.
ДНК нередко сравнивают с чертежами — и, одновременно, инструкциями — для изготовления белков. Развивая эту инженерно-производственную аналогию, можно сказать, что, если ДНК — «полный набор чертежей-инструкций для изготовления белков, находящийся на хранении в сейфе директора завода», то мРНК — «временная рабочая копия чертежа-инструкции для отдельной детали, выдаваемая в сборочный цех». Следует отметить, что ДНК содержат не детальный образ взрослого организма, а больше похожа на «рецепт» по его изготовлению, который применяется в зависимости от сложившихся текущих условий в ходе экспрессии генов — какие-то из полного набора инструкций используются, а какие-то — нет.
История открытия
К середине XX века были накоплены научные данные, которые позволили заключить, что структура белков кодируется участками ДНК — генами[2]. Однако непосредственный механизм кодирования не был установлен.
Работы Ж. Браше (1944) и Т. Касперссона (1947) показали, что клетки, активно синтезирующие белок, содержат большое количество РНК в цитоплазме. Впоследствии выяснилось, что это относится главным образом к рибосомальной РНК, а не к мРНК, количество которой в клетке относительно невелико. Тем не менее, это наблюдение связывало между собой ДНК, РНК и белок и, вероятно, сыграло роль в предположении о возможной роли РНК как посредника, способного переносить информацию от ДНК в ядре к аппарату биосинтеза белка в цитоплазме[3].
В это же время были открыты рибосомы — рибонуклеопротеидные частицы, синтезирующие белок. Было сделано предположение о том, что гены транскрибируются в РНК рибосом, которые и служат матрицами для синтеза белка[4]. Однако в 1956—1958 годах А. Белозерский и А. Спирин, проведя сравнительный анализ нуклеотидного состава ДНК и РНК ряда микроорганизмов, показали, что, при больших вариациях в составе ДНК, РНК разных видов были довольно похожи[5]. Это указывало на то, что основная масса клеточной РНК (рРНК) не отражает нуклеотидный состав ДНК данного организма и не может служить матрицей для синтеза белков. В то же время авторам удалось наблюдать слабую положительную корреляцию между составом ДНК и РНК при больших различиях между видами. Это позволило им предположить, что в клетке, помимо рРНК, существует ещё одна небольшая фракция РНК, которая может быть посредником при экспрессии генов.
Независимо Э. Волкин и Л. Астрачан пришли к сходным выводам: они обнаружили, что при заражении бактериальных клеток бактериофагом Т2 они полностью переключаются на синтез белков вируса. В то время как большая часть РНК клетки-хозяина остаётся неизменной, после заражения синтезируется небольшое количество короткоживущей РНК, сходной по нуклеотидному составу с ДНК фага[6][7].
В 1961 году несколькими группами исследователей было прямо доказано существование короткоживущего РНК-посредника, близкого по структуре к генам в ДНК, который служит матрицей для синтеза белка, связываясь с рибосомами[8][9].
«Жизненный цикл»
Жизненный цикл молекулы мРНК начинается её «считыванием» с матрицы ДНК (транскрипция) и завершается её деградацией до отдельных нуклеотидов. Молекула мРНК в течение своей жизни может подвергаться различным модификациям перед синтезом белка (трансляцией). Эукариотические молекулы мРНК часто требуют сложной обработки и транспортировки из ядра — места синтеза мРНК, на рибосомы, где происходит трансляция, в то время как прокариотические молекулы мРНК этого не требуют и синтез РНК у них сопряжён с синтезом белка[10].
Транскрипция
Транскрипцией называют процесс копирования генетической информации с ДНК на РНК, в частности на мРНК. Транскрипция осуществляется ферментом РНК-полимеразой, строящей, согласно принципу комплементарности, копию участка ДНК на основании одной из цепей двойной спирали. Этот процесс как у эукариот, так и у прокариот организован одинаково. Основное различие между про- и эукариотами состоит в том, что у эукариот РНК-полимераза во время транскрипции ассоциируется с мРНК-обрабатывающими ферментами, поэтому у них обработка мРНК и транскрипция могут проходить одновременно. Короткоживущие необработанные или частично обработанные продукты транскрипции называются пре-мРНК; после полной обработки — зрелая мРНК.
Созревание эукариотической мРНК
В то время как мРНК прокариот (бактерий и архей), за редкими исключениями, сразу готовы к трансляции и не требуют специальной обработки, эукариотические пре-мРНК подвергаются интенсивным модификациям. Так, одновременно с транскрипцией происходит добавление на 5'-конец молекулы РНК специального модифицированного нуклеотида (кэпа), удаление определённых участков РНК (сплайсинг), а также добавление на 3'-конец адениновых нуклеотидов (так называемый полиадениновый, или поли(А)-, хвост)[11]. Обычно эти посттранскрипционные изменения мРНК эукариот обозначают термином «процессинг мРНК».
Кэпирование является первым этапом процессинга мРНК. Оно осуществляется, когда синтезируемый транскрипт достигает длины 25—30 нуклеотидов[12]. Сразу после присоединения кэпа к 5'-концу транскрипта с ним связывается кэп-связывающий комплекс CBC (англ. cap binding complex), который остаётся связанным с мРНК до завершения процессинга и важен для всех последующих его этапов[13]. В процессе сплайсинга из пре-мРНК удаляются не кодирующие белок последовательности — интроны. Полиаденилирование необходимо для транспорта большинства мРНК в цитоплазму и защищает молекулы мРНК от быстрой деградации (увеличивает время их полужизни). Лишённые поли(А)-участка молекулы мРНК (например, вирусные) быстро разрушаются в цитоплазме клеток эукариот рибонуклеазами.
После завершения всех стадий процессинга мРНК проходит проверку на отсутствие преждевременных стоп-кодонов, после чего она становится полноценной матрицей для трансляции[14]. В цитоплазме кэп узнаётся факторами инициации, белками, отвечающими за присоединение к мРНК рибосомы, полиадениновый хвост связывается со специальным поли(А)-связывающим белком PABP1.
Сплайсинг

Сплайсинг — процесс, в котором из пре-мРНК удаляются участки, не кодирующие белок, называемые интронами; последовательности, которые остаются, несут информацию о структуре белка и называются экзонами. Иногда продукты сплайсинга пре-мРНК могут быть соединены разными способами, позволяя одному гену кодировать несколько белков. Этот процесс называется альтернативным сплайсингом. Сплайсинг обычно производится РНК-белковым комплексом, который называется сплайсосома, но некоторые молекулы мРНК также могут катализировать сплайсинг без участия белков (см. рибозимы)[15].
Транспорт
Другое различие между эукариотами и прокариотами — транспорт мРНК. Из-за того, что эукариотические транскрипция и трансляция пространственно разделены, эукариотические мРНК должны быть выведены из ядра в цитоплазму[16]. Зрелые мРНК распознаются по наличию модификаций и покидают ядро через ядерные поры, в цитоплазме мРНК образует нуклеопротеидные комплексы — информосомы, в составе которых транспортируется к рибосомам. Многие мРНК содержат сигналы, которые определяют их локализацию[17]. В нейронах мРНК должна транспортироваться из тела нейронов в дендриты, где трансляция происходит в ответ на внешние раздражители[18].
Экспорт мРНК осуществляется при участии комплекса транспортных факторов Mex67—Mtr2 (у дрожжей) или TAP—p15 (у многоклеточных)[19]. Однако этот комплекс связывает мРНК не напрямую, а через адаптерный белок Yra1 (у дрожжей) или ALY/REF (у многоклеточных), который является одной из субъединиц белкового комплекса TREX. В свою очередь, TREX привлекается в комплекс с мРНК за счёт прямого взаимодействия ALY/REF с CBC80 субъединицей кэп-связывающего комплекса[20]. Такой механизм обеспечивает присоединение транспортного комплекса близко к 5'-концу мРНК и соответствующую направленность её транспорта, 5'-концом в сторону цитоплазмы.
Метилирование
мРНК эукариот подвергаются посттранскрипционному метилированию. Наиболее распространённой модификацией является метилирование остатков аденозина по положению N6 с образованием N6-метиладенозина (m6A). Этот процесс катализируют ферменты N6-аденозинметилтрансферазы, которые распознают остатки аденозина в консенсусных последовательностях GAC (70 % случаев) и AAC (30 % случаев). Соответствующие деметилазы катализируют обратный процесс деметилирования. Учитывая обратимость и динамичность процесса метилирования мРНК, а также повышенную концентрацию m6A в длинных экзонах и вокруг стоп-кодонов, предполагают, что метилирование мРНК выполняет регуляторную функцию[21].
Трансляция
Поскольку прокариотическая мРНК не нуждается в обработке и транспортировке, трансляция рибосомой может начаться немедленно после транскрипции. Следовательно, можно сказать, что трансляция у прокариот совмещена с транскрипцией и происходит ко-транскрипционно.
Эукариотическая мРНК должна быть обработана и доставлена из ядра в цитоплазму, и только тогда может быть транслирована рибосомой. Трансляция может происходить как на рибосомах, находящихся в цитоплазме в свободном виде, так и на рибосомах, ассоциированных со стенками эндоплазматического ретикулума. Таким образом, у эукариот трансляция не совмещена напрямую с транскрипцией.
Регуляция трансляции
Так как у прокариот транскрипция совмещена с трансляцией, прокариотическая клетка может быстро реагировать на изменения в окружающей среде путём синтеза новых белков, то есть регуляция происходит, в основном, на уровне транскрипции. У эукариот из-за необходимости процессинга и транспорта мРНК ответ на внешние стимулы занимает больше времени. Поэтому их синтез белка интенсивно регулируется на посттранскрипционном уровне. Не всякая зрелая мРНК транслируется, поскольку в клетке существуют механизмы регуляции экспрессии белков на посттранскрипционном уровне, например, РНК-интерференция.
Некоторые мРНК в действительности содержат два тандемных терминаторных кодона (стоп-кодона) — часто это кодоны различного типа на конце кодирующей последовательности[22].
Строение зрелой мРНК

Зрелая мРНК состоит из нескольких участков, различающихся по функциям: «5'-кэп», 5'-нетранслируемая область, кодирующая (транслируемая) область, 3'-нетранслируемая область и 3'-полиадениновый «хвост».
5'-Кэп
5'-кэп (от англ. cap — шапочка) — модифицированный гуанозиновый нуклеотид, который добавляется на 5'- (передний) конец незрелой мРНК. Эта модификация очень важна для узнавания мРНК при инициации трансляции, а также для защиты от 5'-нуклеаз — ферментов, разрушающих цепи нуклеиновых кислот с незащищённым 5'-концом.
Кодирующие области
Кодирующие области состоят из кодонов — следующих непосредственно друг за другом последовательностей из трёх нуклеотидов, каждая из которых соответствует в генетическом коде определённой аминокислоте или началу и концу синтеза белка. Кодирующие области начинаются со старт-кодона и заканчиваются одним из трёх стоп-кодонов. Считывание последовательности кодонов и сборка на её основе последовательности аминокислот синтезируемой молекулы белка осуществляется рибосомами при участии транспортных РНК в процессе трансляции. В дополнение к кодированию белков, части кодирующих областей могут служить управляющими последовательностями. Например, вторичная структура РНК в некоторых случаях определяет результат трансляции.
Моноцистронная и полицистронная мРНК
мРНК называют моноцистронной, если она содержит информацию, необходимую для трансляции только одного белка (один цистрон). Полицистронная мРНК кодирует несколько белков. Гены (цистроны) в такой мРНК разделены интергенными, некодирующими последовательностями. Полицистронные мРНК характерны для прокариот и вирусов, у эукариот большая часть мРНК является моноцистронной[23][24][25].
Нетранслируемые области
Нетранслируемые области — участки РНК, расположенные до старт-кодона и после стоп-кодона, которые не кодируют белок. Они называются 5'-нетранслируемая область и 3'-нетранслируемая область, соответственно. Эти области транскрибируются в составе того же самого транскрипта, что и кодирующий участок. Нетранслируемые области имеют несколько функций в жизненном цикле мРНК, включая регуляцию стабильности мРНК, локализации мРНК и эффективности трансляции. Стабильность мРНК может контролироваться 5'- и/или 3'-областью из-за различной чувствительности к ферментам, которые отвечают за деградацию РНК — РНКазам и регуляторным белкам, которые убыстряют или замедляют деградацию[26].
3'-полиадениновый хвост
Длинная (часто несколько сотен нуклеотидов) последовательность адениновых оснований, которая присутствует на 3'-«хвосте» мРНК эукариот, синтезируется ферментом полиаденилатполимеразой. У высших эукариот поли(А)-хвост добавляется к транскрибированной РНК, которая содержит специфическую последовательность, AAUAAA. Важность этой последовательности можно увидеть на примере мутации в гене человеческого 2-глобина, которая изменяет AAUAAA на AAUAAG, что приводит к недостаточному количеству глобина в организме[27].
Вторичная структура
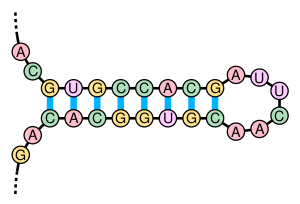

Кроме первичной структуры (последовательности нуклеотидов), мРНК обладает вторичной структурой. В отличие от ДНК — вторичная структура которой основана на межмолекулярных взаимодействиях (двойная спираль ДНК образована двумя линейными молекулами, соединенными друг с другом по всей длине водородными связями), — вторичная структура мРНК основана на внутримолекулярных взаимодействиях (линейная молекула «складывается», и водородные связи возникают между разными участками одной и той же молекулы).
Примерами вторичной структуры могут служить стебель, петля и псевдоузел.[28]
Вторичные структуры в мРНК служат для регуляции трансляции. Например, вставка в белки необычных аминокислот, селенометионина и пирролизина, зависит от стебля-петли, расположенной в 3'-нетранслируемой области. Псевдоузлы служат для программированного изменения рамки считывания генов. Также вторичная структура служит для замедления деградации определённых мРНК[29][30]
В вирусных мРНК сложные вторичные структуры (IRES) направляют трансляцию, не зависящую от узнавания кэпа и факторов инициации трансляции (см. «Инициация трансляции»).
Разрушение
Различные мРНК имеют различную продолжительность жизни (стабильность). В клетках бактерий молекула мРНК может существовать от нескольких секунд до более чем часа, а в клетках млекопитающих — от нескольких минут до нескольких дней. Чем больше стабильность мРНК, тем больше белка может быть синтезировано с данной молекулы. Ограниченное время жизни мРНК клетки позволяет быстро изменять синтез белка в ответ на изменяющиеся потребности клетки. По прошествии некоторого времени, определяемого её нуклеотидной последовательностью, — в частности, длиной полиаденинового участка на 3'-конце молекулы, — мРНК разрушается на составляющие её нуклеотиды с участием РНКаз. К настоящему времени известно много механизмов деградации мРНК, некоторые из которых описаны ниже.
Деградация мРНК у прокариот
У прокариот стабильность мРНК намного меньше, чем у эукариот. Деградация мРНК в клетках прокариот происходит под действием комбинации рибонуклеаз, в том числе эндонуклеаз, 3'-экзонуклеаз и 5'-экзонуклеаз. В некоторых случаях малые молекулы РНК длиной от десятков до сотен нуклеотидов могут стимулировать деградацию мРНК, комплементарно спариваясь с соответствующими последовательностями в мРНК и содействуя рибонуклеазам[31][32] . В 2008 году было показано, что бактерии имеют нечто вроде кэпа — трифосфат на 5'-конце[33]. Удаление двух фосфатов оставляет монофосфат на 5'-конце, в результате чего мРНК расщепляется эндонуклеазой РНКаза E.
У эукариот
Как правило, разрушение начинается с удаления кэпа на 5'-конце, полиаденинового хвоста на 3'-конце, и затем нуклеазы одновременно разрушают мРНК в направлениях 5' ->3' и 3' ->5'. мРНК, в которой сигнал завершения синтеза белка, стоп-кодон, в результате ошибки транскрипции находится в середине кодирующей последовательности, подвержена особой быстрой форме деградации, НМД.
Методы определения
В последнее время разработаны очень чувствительные методы, позволяющие проанализировать «транскриптом» из образцов размером в 50—100 клеток[34][35][36].
См. также
Литература
- ↑ Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, Peter Walter. Molecular Biology of the Cell. — 5. — Garland Science, 2008. — 1392 с. — ISBN 0815341059.
- ↑ Ичас М. Биологический код. — Москва: Мир, 1971.
- ↑ Crick F. H. The genetic code - yesterday, today, and tomorrow // Cold Spring Harb. Symp. Quant. Biol.. — 1966. — Т. 31. — С. 1—9. — PMID 5237190.
- ↑ Спирин А. С. Глава II. Информационная РНК и генетический код // Молекулярная биология. Структура рибосомы и биосинтез белка. — Москва: Высшая школа, 1986. — С. 9—11.
- ↑ Belozersky A. N., Spirin A. S. A correlation between the compositions of deoxyribonucleic and ribonucleic acids (англ.) // Nature. — 1958. — Vol. 182, iss. 4628. — P. 111—112. — PMID 13566202.
- ↑ Volkin E., Astrachan L. Intracellular distribution of labeled ribonucleic acid after phage infection of Escherichia coli // Virology. — 1956. — Т. 2, вып. 4. — С. 433—437. — PMID 13352773.
- ↑ Volkin E., Astrachan L. Phosphorus incorporation in Escherichia coli ribo-nucleic acid after infection with bacteriophage T2 // Virology. — 1956. — Т. 2, вып. 2. — С. 149—161. — PMID 13312220.
- ↑ Brenner S., Jacob F., Meselson M. An unstable intermediate carrying information from genes to ribosomes for protein synthesis (англ.) // Nature. — 1961. — Vol. 190. — P. 576—581. — PMID 20446365.
- ↑ Gros F., Hiatt H., Gilbert W., Kurland C. G., Risebrough R. W., Watson J. D. Unstable ribonucleic acid revealed by pulse labelling of Escherichia coli (англ.) // Nature. — 1961. — Vol. 190. — P. 581—585. — PMID 13708983.
- ↑ Alberts, Bruce; Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walters. Molecular Biology of the Cell; Fourth Edition (англ.). — New York and London: Garland Science[англ.], 2002.
- ↑ Moore M. J., Proudfoot N. J. Pre-mRNA processing reaches back to transcription and ahead to translation (англ.) // Cell : journal. — Cell Press, 2009. — Vol. 20. — P. 688—700. — PMID 19239889.
- ↑ Rasmussen E. B., Lis JT. In vivo transcriptional pausing and cap formation on three Drosophila heat shock genes (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 1993. — Vol. 90. — P. 7923—7927. — PMID 8367444.
- ↑ Topisirovic I., Svitkin Y. V., Sonenberg N., Shatkin A. J. Cap and cap-binding proteins in the control of gene expression (англ.) // Wiley Interdiscip Rev RNA : journal. — 2011. — Vol. 2, no. 2. — P. 277—298. — doi:10.1002/wrna.52. — PMID 21957010.
- ↑ Maquat L. E. Nonsense-mediated mRNA decay: splicing, translation and mRNP dynamics (англ.) // Nat. Rev. Mol. Cell Biol. : journal. — 2004. — Vol. 5, no. 2. — P. 89—99. — doi:10.1038/nrm1310. — PMID 15040442.
- ↑ Johnston W., Unrau P., Lawrence M., Glasner M., Bartel D. RNA-catalyzed RNA polymerization: accurate and general RNA-templated primer extension (англ.) // Science : journal. — 2001. — Vol. 292, no. 5520. — P. 1319—1325. — PMID 11358999. Архивировано 27 февраля 2012 года.
- ↑ Paquin N., Chartrand P. Local regulation of mRNA translation: new insights from the bud (англ.) // Trends Cell Biol[англ.] : journal. — 2008. — Vol. 18. — P. 105—111.
- ↑ Ainger, Kevin; Avossa, Daniela; Diana, Amy S. & Barry, Christopher (1997), Transport and Localization Elements in Myelin Basic Protein mRNA, The Journal of Cell Biology Т. 138 (5): 1077–1087, PMID 9281585, doi:10.1083/jcb.138.5.1077, <http://www.jcb.org/cgi/content/full/138/5/1077> Архивная копия от 28 ноября 2007 на Wayback Machine
- ↑ Job, C. & Eberwine, J. (1912), Localization and translation of mRNA in dendrites and axons, Nat Rev Neurosci Т. 2001 (12): 889–98, PMID 11733796, doi:10.1038/35104069, <https://www.ncbi.nlm.nih.gov/pubmed/11733796> Архивная копия от 3 октября 2016 на Wayback Machine
- ↑ Köhler A., Hurt E. Exporting RNA from the nucleus to the cytoplasm (англ.) // Nat. Rev. Mol. Cell Biol. : journal. — 2007. — Vol. 8, no. 10. — P. 761—773. — doi:10.1038/nrm2255. — PMID 17786152.
- ↑ Cheng H., Dufu K., Lee C. S., Hsu J. L., Dias A., Reed R. Human mRNA export machinery recruited to the 5' end of mRNA (англ.) // Cell : journal. — Cell Press, 2006. — Vol. 127, no. 7. — P. 1389—1400. — doi:10.1016/j.cell.2006.10.044. — PMID 17190602.
- ↑ Wang X., Lu Z., Gomez A., Hon G. C., Yue Y., Han D., Fu Y., Parisien M., Dai Q., Jia G., Ren B., Pan T., He C. {{{заглавие}}} (англ.) // Nature. — 2014. — Vol. 505, iss. 7481. — P. 117—120. — doi:10.1038/nature12730. — PMID 24284625.
- ↑ Айала Ф. Д. Современная генетика. 1987.
- ↑ Poyry, T.,Kaminski, A., Jackson R. What determines whertehr mammalian ribosomes resume scanning after translation of a short upstream open reading frame (англ.) // Genes and Development : journal. — 2004. — Vol. 18. — P. 62—75.
- ↑ Kozak, M. (1983), Comparison of initiation of protein synthesis in procaryotes, eucaryotes, and organelles, Microbiological Reviews Т. 47 (1): 1–45, PMID 6343825, <http://www.pubmedcentral.nih.gov/picrender.fcgi?artid=281560&blobtype=pdf>. Проверено 12 августа 2006.
- ↑ Niehrs C, Pollet N (1999), Synexpression groups in eukaryotes, Nature Т. 402 (6761): 483–7, PMID 10591207, DOI 10.1038/990025
- ↑ Kozak, M. Comparison of initiation of protein synthesis in procaryotes, eucaryotes, and organelles (англ.) // Microbiology and Molecular Biology Reviews[англ.] : journal. — American Society for Microbiology[англ.], 1983. — Vol. 47, no. 1. — P. 1—45. — PMID 15680349.
- ↑ Shaw, G. and Kamen, R. A conserved AU sequence from the 3' untranslated region of GM-CSF mRNA mediates selective mRNA degradation (англ.) // Cell : journal. — Cell Press, 1986. — Vol. 46, no. 5. — P. 659—667. — PMID 15680349.
- ↑ Компьютерный анализ процессов структурообразования нуклеиновых кислот // Математическое моделирование. — М., 2013. — Т. 25, № 4. — С. 126–134.
- ↑ Shabalina SA, Ogurtsov AY, Spiridonov NA (2006), A periodic pattern of mRNA secondary structure created by the genetic code, Nucleic Acids Res. Т. 34 (8): 2428–37, PMID 16682450, DOI 10.1093/nar/gkl287
- ↑ Katz L, Burge CB (2003), Widespread selection for local RNA secondary structure in coding regions of bacterial genes, Genome Res. Т. 13 (9): 2042–51, PMID 12952875, DOI 10.1101/gr.1257503
- ↑ Vogel J., Wagner E. G. Target identification of small noncoding RNAs in bacteria (англ.) // Curr. Opin. Microbiol. : journal. — 2007. — June (vol. 10, no. 3). — P. 262—270. — doi:10.1016/j.mib.2007.06.001. — PMID 17574901.
- ↑ Viegas S. C., Arraiano C. M. Regulating the regulators: How ribonucleases dictate the rules in the control of small non-coding RNAs (англ.) // RNA Biol[англ.] : journal. — 2008. — Vol. 5, no. 4. — P. 230—243. — PMID 18981732.
- ↑ Deana, Atilio; Celesnik, Helena & Belasco, Joel G. (2008), The bacterial enzyme RppH triggers messenger RNA degradation by 5' pyrophosphate removal, Nature Т. 451 (7176): 355–8, PMID 18202662, doi:10.1038/nature06475, <http://www.nature.com/nature/journal/v451/n7176/abs/nature06475.html> Архивная копия от 21 января 2008 на Wayback Machine
- ↑ Bhargava, V., Ko, P., Willems, E., Mercola, M., & Subramaniam, S. (2013)Quantitative Transcriptomics using Designed Primer-based Amplification Архивная копия от 27 октября 2013 на Wayback Machine. Scientific reports, 3, Article number: 1740 doi:10.1038/srep01740
- ↑ Tilgner, H., Raha, D., Habegger, L., Mohiuddin, M., Gerstein, M., & Snyder, M. (2013). Accurate Identification and Analysis of Human mRNA Isoforms Using Deep Long Read Sequencing. G3: Genes| Genomes| Genetics, 3(3), 387—397.doi: 10.1534/g3.112.004812
- ↑ Drewe, P., Stegle, O., Hartmann, L., et al. & Rätsch, G. (2013). Accurate detection of differential RNA processing. Nucleic acids research, 41(10), 5189-5198 doi: 10.1093/nar/gkt211
Ссылки
- Жизненный цикл мРНК Архивная копия от 27 сентября 2007 на Wayback Machine Флэш анимация
- База данных модификаций РНК (англ.)
- База данных старт- и стоп-кодонов, нетранслируемых последовательностей (англ.)
- Сайт, содержащий список программ по предсказанию свойств молекул РНК [1] (англ.)