Orthornavirae
| Orthornavirae | |
|---|---|
 По часовой стрелке сверху слева: ПЭМ птичьего коронавируса, вируса полиомиелита, бактериофага Qβ, эболавируса, вируса табачной мозаики, вируса гриппа А, ротавируса, вирус везикулярного стоматита. Центр: филогенетическое древо белка общей репликации RdRp. | |
| Научная классификация | |
| Международное научное название | |
| Orthornavirae | |
| Типы и классы | |
Orthornavirae — царство вирусов, геномы которых состоят из рибонуклеиновой кислоты (РНК) и кодируют РНК-зависимую РНК-полимеразу (RdRp). RdRp используется для транскрипции генома вирусной РНК в информационную РНК (мРНК) и для репликации генома. Вирусы этого царства также имеют ряд общих характеристик, связанных с эволюцией, включая высокую скорость генетических мутаций, рекомбинаций и реассортации.
Вирусы Orthornavirae принадлежат к реалму Riboviria. Они произошли от общего предка, который мог быть невирусной молекулой, кодирующей обратную транскриптазу вместо RdRp для репликации. Царство подразделяется на пять типов, которые разделяют вирусы-члены на основе их типа генома, диапазона хозяев и генетического сходства. Включаются вирусы с тремя типами генома: РНК-вирусы с положительной цепью, РНК-вирусы с отрицательной цепью и двухцепочечные РНК-вирусы .
Многие из наиболее широко известных вирусных заболеваний вызываются РНК-вирусами в королевстве, включая коронавирусы, вирус Эбола, вирусы гриппа, вирус кори и вирус бешенства. Первый обнаруженный вирус, вирус табачной мозаики, относится к королевству. В современной истории РНК-вирусы, кодирующие RdRp, вызывали многочисленные вспышки болезней и поражали многие экономически важные сельскохозяйственные культуры. Большинство эукариотических вирусов, включая большинство вирусов человека, животных и растений, представляют собой РНК-вирусы, кодирующие RdRp. Напротив, в королевстве относительно мало прокариотических вирусов.
Этимология
Первая часть Orthornavirae происходит от греческого ὀρθός [orthós], что означает «прямой», средняя часть, rna, относится к РНК, а -virae — это суффикс, используемый для царств вирусов[2].
Характеристика
Структура
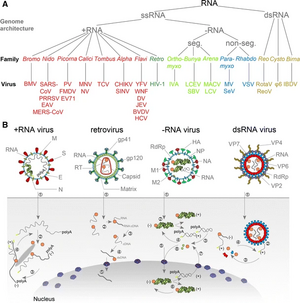
РНК-вирусы Orthornavirae обычно не кодируют множество белков. Большинство одноцепочечных (+ssRNA) вирусов с положительным смыслом и некоторые двухцепочечные РНК-вирусы (dsRNA) кодируют основной белок капсида, который имеет одиночную желеобразную укладку, так называемую потому что складчатая структура белка содержит структуру, напоминающую рулет из желе[3]. Многие также обладают оболочкой из липидной мембраны, которая обычно окружает капсид. В частности, вирусная оболочка почти универсальна среди одноцепочечных (-ssRNA) вирусов с отрицательным смыслом[4][5].
Геном
Вирусы Orthornavirae имеют три различных типа геномов: dsRNA, +ssRNA и -ssRNA. Одноцепочечные РНК-вирусы имеют либо положительную, либо отрицательную смысловую цепь, а вирусы dsRNA имеют обе. Эта структура генома важна с точки зрения транскрипции для синтеза вирусной мРНК, а также репликации генома, которые осуществляются вирусным ферментом РНК-зависимой РНК-полимеразой (RdRp), также называемой РНК-репликазой[2][3].
Репликация и транскрипция
РНК-вирусы с положительной цепью
РНК-вирусы с положительной цепью имеют геномы, которые могут функционировать как мРНК, поэтому транскрипция не требуется. Однако +ssRNA будут продуцировать формы dsRNA как часть процесса репликации их геномов. Из dsRNA синтезируются дополнительные положительные цепи, которые можно использовать в качестве мРНК или геномов для потомства. Поскольку вирусы +ssRNA создают промежуточные формы dsRNA, им приходится избегать иммунной системы хозяина, чтобы размножаться. Вирусы +ssRNA достигают этого путем репликации в ассоциированных с мембраной везикулах, которые используются в качестве фабрик репликации. Для многих вирусов +ssRNA субгеномные части генома будут транскрибироваться для трансляции определённых белков, тогда как другие будут транскрибировать полипротеин, который расщепляется с образованием отдельных белков[6][7].
РНК-вирусы с отрицательной цепью
РНК-вирусы с отрицательной цепью имеют геномы, которые функционируют как матрицы, из которых мРНК может быть синтезирована непосредственно с помощью RdRp[8]. Репликация — это тот же процесс, но выполняемый на антигеноме положительного смысла, во время которого RdRp игнорирует все сигналы транскрипции, так что может быть синтезирован полный геном -ssRNA[9]. Вирусы -ssRNA различаются между вирусами, которые инициируют транскрипцию с помощью RdRp, создавая кэп на 5'-конце (обычно произносится как «5 прайм конец») генома, или отрывая кэп от мРНК хозяина и прикрепляя его к вирусу. РНК[10]. У многих вирусов -ssRNA в конце транскрипции RdRp «заикается» на урациле в геноме, синтезируя сотни аденинов подряд как часть создания полиаденилированного хвоста для мРНК[11]. Некоторые вирусы -ssRNA по существу амбисенсны и имеют белки, кодируемые как положительной, так и отрицательной цепью, поэтому мРНК синтезируется непосредственно из генома и из комплементарной цепи[12].
Двухцепочечные РНК-вирусы
Для вирусов dsRNA RdRp транскрибирует мРНК, используя отрицательную цепь в качестве матрицы. Положительные цепи также можно использовать в качестве матриц для синтеза отрицательных цепей для конструирования геномной dsRNA. dsRNA не является молекулой, продуцируемой клетками, поэтому клеточная жизнь выработала механизмы для обнаружения и инактивации вирусной dsRNA. Чтобы противостоять этому, вирусы dsRNA обычно сохраняют свои геномы внутри вирусного капсида, чтобы уклониться от иммунной системы хозяина[13].
Эволюция
РНК-вирусы Orthornavirae подвержены высокой частоте генетических мутаций, потому что RdRp склонен к ошибкам при репликации, поскольку в нём обычно отсутствуют механизмы корректуры для исправления ошибок[note 1]. На мутации РНК-вирусов часто влияют факторы хозяина, такие как дцРНК-зависимые аденозиндеаминазы, которые редактируют вирусные геномы, заменяя аденозины на инозины[14][15]. Мутации в генах, которые необходимы для репликации, приводят к уменьшению числа потомков, поэтому вирусные геномы обычно содержат высококонсервативные последовательности с относительно небольшим количеством мутаций[16].
Многие РНК-вирусы, кодирующие RdRp, также испытывают высокую скорость генетической рекомбинации, хотя скорость рекомбинации значительно различается: более низкая скорость у вирусов -ssRNA и более высокая скорость у вирусов dsRNA и +ssRNA. Существует два типа рекомбинации: рекомбинация выбора копии и реассортация. Рекомбинация выбора копии происходит, когда RdRp переключает матрицы во время синтеза, не высвобождая предшествующую, вновь созданную цепь РНК, которая генерирует геном смешанного происхождения. Реассортация, которая ограничена вирусами с сегментированными геномами, имеет сегменты из разных геномов, упакованные в один вирион или вирусную частицу, которая также производит гибридное потомство[14][17].
Для рекомбинации некоторые сегментированные вирусы упаковывают свои геномы в несколько вирионов, в результате чего геномы представляют собой случайные смеси родителей, тогда как для тех, которые упакованы в один вирион, обычно отдельные сегменты меняются местами. Обе формы рекомбинации могут возникать только в том случае, если в клетке присутствует более одного вируса, и чем больше аллелей присутствует, тем более вероятна рекомбинация. Ключевое различие между рекомбинацией выбора копии и реассортацией состоит в том, что рекомбинация выбора копии может происходить в любом месте генома, тогда как реассортация меняет местами полностью реплицированные сегменты. Следовательно, рекомбинация выбора копии может продуцировать нефункциональные вирусные белки, тогда как реассортация не может[14][17][18].
Скорость мутаций вируса связана со скоростью генетических рекомбинаций. Более высокие скорости мутаций увеличивают количество как полезных, так и неблагоприятных мутаций, тогда как более высокие скорости рекомбинации позволяют отделить полезные мутации от вредных. Следовательно, более высокие скорости мутаций и рекомбинаций до определённого момента улучшают способность вирусов к адаптации[14][19]. Известные примеры этого включают рекомбинацию, которая способствует межвидовой передаче вирусов гриппа, что привело к многочисленным пандемиям, а также появление штаммов гриппа с лекарственной устойчивостью через мутации, которые были реассортированы[20].
Филогенетика

Точное происхождение Orthornavirae точно не установлено, но вирусный RdRp демонстрирует связь с ферментами обратной транскриптазы (RT) интронов группы II, которые кодируют RT и ретротранспозоны, последние из которых представляют собой самореплицирующиеся последовательности ДНК, которые интегрируются в другие частей одной и той же молекулы ДНК. Внутри королевства вирусы +ssRNA, вероятно, являются самой старой линией, вирусы dsRNA, по-видимому, неоднократно возникали из вирусов +ssRNA, а вирусы -ssRNA, в свою очередь, по-видимому, связаны с реовирусами, которые являются вирусами dsRNA[2][21].
Классификация
РНК-вирусы, кодирующие RdRp, отнесены к царству Orthornavirae, которое содержит пять типов и несколько таксонов, не отнесенных к типу из-за отсутствия информации. Пять типов разделены на основе типов генома, диапазонов хозяев и генетического сходства вирусов-членов[2][22].
- Тип: Duplornaviricota, который содержит вирусы dsRNA, инфицирующие прокариоты и эукариоты, которые не кластеризуются с членами Pisuviricota и которые кодируют капсид, состоящий из 60 гомо- или гетеродимеров капсидных белков, организованных на решетке с псевдосимметрией T=2.
- Тип: Kitrinoviricota, содержащий вирусы +ssRNA, инфицирующие эукариот и не группирующиеся с представителями Pisuviricota.
- Тип: Lenarviricota, содержащий вирусы +ssRNA, инфицирующие прокариоты и эукариоты и не группирующиеся с представителями Kitrinoviricota.
- Тип: Negarnaviricota, который содержит все вирусы -ssRNA[note 2].
- Тип: Pisuviricota, который содержит вирусы +ssRNA и dsRNA, инфицирующие эукариоты и не кластеризующиеся с другими типами.
Неназначенные таксоны перечислены ниже (-viridae обозначает семейство, а -virus обозначает род)[2][22].
- Birnaviridae
- Permutotetraviridae
- Botybirnavirus
Королевство состоит из трех групп в системе классификации Балтимора, которая объединяет вирусы на основе их способа синтеза мРНК и часто используется вместе со стандартной таксономией вирусов, основанной на истории эволюции. Эти три группы представляют собой группу III: вирусы dsRNA, группу IV: вирусы + ssRNA и группу V: вирусы -ssRNA[2][23].
Болезни
РНК-вирусы связаны с широким спектром заболеваний, включая многие из наиболее широко известных вирусных заболеваний. Известные болезнетворные вирусы Orthornavirae включают:[22]
- Coronaviruses
- Crimean-Congo hemorrhagic fever orthonairovirus
- Dengue virus
- Ebolavirus
- Hantaviruses
- Hepatitis A virus
- Hepatitis C virus
- Hepatitis E virus
- Human orthopneumovirus
- Influenza viruses
- Japanese encephalitis virus
- Lassa mammarenavirus
- Measles morbillivirus
- Mumps orthorubulavirus
- Norovirus
- Poliovirus
- Rabies lyssavirus
- Rhinoviruses
- Rift Valley fever phlebovirus
- Rotavirus
- Rubella virus
- West Nile virus
- Yellow fever virus
- Zika virus
Вирусы животных в Orthornavirae включают орбивирусы, которые вызывают различные заболевания у жвачных животных и лошадей, в том числе вирус катаральной лихорадки, вирус африканской чумы лошадей, вирус энцефалеза лошадей и вирус эпизоотической геморрагической болезни[24]. Вирус везикулярного стоматита вызывает заболевание крупного рогатого скота, лошадей и свиней[25]. Летучие мыши являются переносчиками многих вирусов, включая эболавирусы и генипавирусы, которые также могут вызывать заболевания у людей[26]. Аналогично, вирусы членистоногих родов Flavivirus и Phlebovirus многочисленны и часто передаются человеку[27][28]. Коронавирусы и вирусы гриппа вызывают заболевания у различных позвоночных, включая летучих мышей, птиц и свиней[29][30].
Вирусы растений в королевстве многочисленны и поражают многие экономически важные культуры. По оценкам, вирус пятнистого увядания томатов ежегодно наносит ущерб на сумму более 1 миллиарда долларов США, поражая более 800 видов растений, включая хризантему, салат, арахис, перец и помидоры. Вирус мозаики огурца поражает более 1200 видов растений и также вызывает значительные потери урожая. Вирус Y картофеля вызывает значительное снижение урожайности и качества перца, картофеля, табака и томатов, а вирус оспы сливы является наиболее важным вирусом среди косточковых культур. Вирус мозаики костреца, хотя и не вызывает значительных экономических потерь, встречается на большей части мира и в основном поражает травы, в том числе злаки[31][32].
История
Заболевания, вызываемые РНК-содержащими вирусами Orthornavirae, были известны на протяжении большей части истории, но их причина была обнаружена только в наше время. В целом, РНК-вирусы были открыты в период крупных достижений в области молекулярной биологии, включая открытие мРНК как непосредственного носителя генетической информации для синтеза белка[33]. Вирус табачной мозаики был обнаружен в 1898 году и стал первым обнаруженным вирусом[34]. Вирусы в королевстве, которые передаются членистоногими, были ключевой мишенью в разработке средств борьбы с переносчиками, которые часто направлены на предотвращение вирусных инфекций[35]. В современной истории многочисленные вспышки заболеваний были вызваны РНК-вирусами, кодирующими RdRp, включая вспышки, вызванные коронавирусами, лихорадкой Эбола и гриппом[36].
Orthornavirae была создана в 2019 году как царство в реалме Riboviria, предназначенное для размещения всех РНК-вирусов, кодирующих RdRp. До 2019 года Riboviria была создана в 2018 году и включала только RdRp-кодирующие РНК-вирусы. В 2019 году Riboviria были расширены за счет включения вирусов с обратной транскрипцией, помещенных в царство Pararnavirae, поэтому Orthornavirae была создана для отделения РНК-вирусов, кодирующих RdRp, от вирусов с обратной транскрипцией[2][37].
Галерея
-
Parotiditis (Paramyxoviridae) -->
Заметки
Использованная литература
- ↑ Таксономия вирусов (англ.) на сайте Международного комитета по таксономии вирусов (ICTV).
- ↑ 2,0 2,1 2,2 2,3 2,4 2,5 2,6 Create a megataxonomic framework, filling all principal taxonomic ranks, for realm Riboviria (англ.). International Committee on Taxonomy of Viruses (ICTV) (18 October 2019).
- ↑ 3,0 3,1 Yuri I. Wolf, Darius Kazlauskas, Jaime Iranzo, Adriana Lucía-Sanz, Jens H. Kuhn. Origins and Evolution of the Global RNA Virome (англ.) // mBio / Vincent R. Racaniello. — 2018-12-21. — Vol. 9, iss. 6. — P. e02329–18. — ISSN 2150-7511 2161-2129, 2150-7511. — doi:10.1128/mBio.02329-18.
- ↑ Viral budding ~ ViralZone. viralzone.expasy.org. Дата обращения: 20 августа 2022.
- ↑ Paula Tennant. Viruses : molecular biology, host interactions, and applications to biotechnology. — London, 2018. — 1 online resource (xvi, 375 pages) с. — ISBN 978-0-12-811194-9, 0-12-811194-1.
- ↑ +RNA virus replication/transcription ~ ViralZone. viralzone.expasy.org. Дата обращения: 20 августа 2022.
- ↑ Subgenomic RNA transcription. ViralZone. Swiss Institute of Bioinformatics. Дата обращения: 6 августа 2020.
- ↑ Negative-stranded RNA virus transcription ~ ViralZone. viralzone.expasy.org. Дата обращения: 20 августа 2022.
- ↑ Negative stranded RNA virus replication. ViralZone. Swiss Institute of Bioinformatics. Дата обращения: 6 августа 2020.
- ↑ Cap snatching. ViralZone. Swiss Institute of Bioinformatics. Дата обращения: 6 августа 2020.
- ↑ Negative-stranded RNA virus polymerase stuttering. ViralZone. Swiss Institute of Bioinformatics. Дата обращения: 6 августа 2020.
- ↑ Ambisense transcription in negative stranded RNA viruses. ViralZone. Swiss Institute of Bioinformatics. Дата обращения: 6 августа 2020.
- ↑ dsRNA replication/transcription ~ ViralZone. viralzone.expasy.org. Дата обращения: 20 августа 2022.
- ↑ 14,0 14,1 14,2 14,3 Rafael Sanjuán, Pilar Domingo-Calap. Mechanisms of viral mutation (англ.) // Cellular and Molecular Life Sciences. — 2016-12. — Vol. 73, iss. 23. — P. 4433–4448. — ISSN 1420-9071 1420-682X, 1420-9071. — doi:10.1007/s00018-016-2299-6.
- ↑ Smith EC (27 April 2017). «The not-so-infinite malleability of RNA viruses: Viral and cellular determinants of RNA virus mutation rates». PLOS Pathog 13 (4): e1006254. doi:10.1371/journal.ppat.1006254. PMID 28448634.
- ↑ (March 2008) «Highly conserved regions of influenza a virus polymerase gene segments are critical for efficient viral RNA packaging». J Virol 82 (5): 2295–2304. doi:10.1128/JVI.02267-07. PMID 18094182.
- ↑ 17,0 17,1 Etienne Simon-Loriere, Edward C. Holmes. Why do RNA viruses recombine? (англ.) // Nature Reviews Microbiology. — 2011-08. — Vol. 9, iss. 8. — P. 617–626. — ISSN 1740-1534 1740-1526, 1740-1534. — doi:10.1038/nrmicro2614.
- ↑ (July 2016) «Reassortment in segmented RNA viruses: mechanisms and outcomes». Nat Rev Microbiol 14 (7): 448–460. doi:10.1038/nrmicro.2016.46. PMID 27211789.
- ↑ (23 November 1999) «Mutation rates among RNA viruses». Proc Natl Acad Sci U S A 96 (24): 13910–13913. doi:10.1073/pnas.96.24.13910. PMID 10570172. .
- ↑ (9 July 2015) «RNA Virus Reassortment: An Evolutionary Mechanism for Host Jumps and Immune Evasion». PLOS Pathog 11 (7): e1004902. doi:10.1371/journal.ppat.1004902. PMID 26158697.
- ↑ Yuri I. Wolf, Darius Kazlauskas, Jaime Iranzo, Adriana Lucía-Sanz, Jens H. Kuhn. Origins and Evolution of the Global RNA Virome (англ.) // mBio / Vincent R. Racaniello. — 2018-12-21. — Vol. 9, iss. 6. — P. e02329–18. — ISSN 2150-7511 2161-2129, 2150-7511. — doi:10.1128/mBio.02329-18.
- ↑ 22,0 22,1 22,2 Current ICTV Taxonomy Release | ICTV. ictv.global. Дата обращения: 20 августа 2022.
- ↑ (27 November 2018) «Origins and Evolution of the Global RNA Virome». mBio 9 (6): e02329-18. doi:10.1128/mBio.02329-18. PMID 30482837.
- ↑ (December 2010) «Re-emergence of bluetongue, African horse sickness, and other orbivirus diseases». Vet Res 41 (6): 35. doi:10.1051/vetres/2010007. PMID 20167199. Проверено 15 August 2020.
- ↑ (11 December 2018) «Vesicular Stomatitis Virus Transmission: A Comparison of Incriminated Vectors». Insects 9 (4): 190. doi:10.3390/insects9040190. PMID 30544935.
- ↑ (February 2019) «Viruses in bats and potential spillover to animals and humans». Curr Opin Virol 34: 79–89. doi:10.1016/j.coviro.2018.12.007. PMID 30665189.
- ↑ (30 April 2017) «Historical Perspectives on Flavivirus Research». Viruses 9 (5): 97. doi:10.3390/v9050097. PMID 28468299.
- ↑ (June 2017) «Rift Valley Fever». Clin Lab Med 37 (2): 285–301. doi:10.1016/j.cll.2017.01.004. PMID 28457351.
- ↑ Coronaviruses. — С. 1–23. — ISBN 978-1-4939-2437-0.
- ↑ (September 2014) «Continuing challenges in influenza». Ann N Y Acad Sci 1323 (1): 115–139. doi:10.1111/nyas.12462. PMID 24891213. .
- ↑ Virus Taxonomy: 2019 Release. talk.ictvonline.org. International Committee on Taxonomy of Viruses. Дата обращения: 6 августа 2020.
- ↑ (December 2011) «Top 10 plant viruses in molecular plant pathology». Mol Plant Pathol 12 (9): 938–954. doi:10.1111/j.1364-3703.2011.00752.x. PMID 22017770.
- ↑ (April 2015) «A short biased history of RNA viruses». RNA 21 (4): 667–669. doi:10.1261/rna.049916.115. PMID 25780183. Проверено 6 August 2020.
- ↑ (29 March 1999) «Milestones in the Research on Tobacco Mosaic Virus». Philos Trans R Soc Lond B Biol Sci 354 (1383): 521–529. doi:10.1098/rstb.1999.0403. PMID 10212931.
- ↑ (16 January 2020) «The Importance of Vector Control for the Control and Elimination of Vector-Borne Diseases». PLOS Negl Trop Dis 14 (1): e0007831. doi:10.1371/journal.pntd.0007831. PMID 31945061.
- ↑ (30 May 2018) «An Evaluation of Emergency Guidelines Issued by the World Health Organization in Response to Four Infectious Disease Outbreaks». PLOS ONE 13 (5): e0198125. doi:10.1371/journal.pone.0198125. PMID 29847593. .
- ↑ Gorbalenya; Krupovic, Mart; Siddell, Stuart; Varsani, Arvind; Kuhn, Jens H. Riboviria: establishing a single taxon that comprises RNA viruses at the basal rank of virus taxonomy (англ.) (docx). International Committee on Taxonomy of Viruses (ICTV) (15 October 2018). Дата обращения: 6 августа 2020.