AutoDock
| AutoDock | |
|---|---|
| Тип | Молекулярное моделирование |
| Разработчик | Scripps Research Institute |
| Написана на | C / C++ |
| Операционная система | Windows, macOS, Linux, Solaris |
| Последняя версия | 4.2.6 (2012-08-04) |
| Состояние | активное |
| Лицензия | GPL2+ (AutoDock 4) / ASL 2.0 (AutoDock Vina) |
| Ссылки | |
| Сайт | autodock.scripps.edu |
AutoDock — пакет программ, предназначенный для автоматизированного молекулярного докинга. В основном применяется для белок-лигандного докинга, в том числе, с учётом подвижных остатков белка. Autodock также используется для «слепого докинга», когда активный центр белка не известен.
О программе
AutoDock — один из пакетов программ, позволяющих предсказывать связывание маленьких молекул с белками известной структуры. Настоящие дистрибутивы AutoDock включают в себя два поколения ПО: AutoDock 4 и AutoDock Vina. AutoDock является свободным программным обеспечением, последняя 4-я версия которого распространяется в соответствии с открытым лицензионным соглашением GNU General Public License, AutoDock Vina доступна под Apache license[1][2].
Начиная с самой первой версии, Autodock представляет собой совокупность 2х программ: Autodock — собственно программа, проводящая докинг и Autogrid — программа, позволяющая рассчитывать сетки потенциалов. Для каждого рецептора (рецептором в докинге называется макромолекула, для которой рассчитываются сетки) достаточно рассчитать сетки потенциалов для каждого типа атомов один раз, что позволяет запускать расчеты для любого лиганда, состоящего из этих атомов (лиганд в докинге это маленькая молекула, для которой возможно изменение положения и конформации)[1].
В 2006 г. AutoDock был наиболее цитируемой программой для докинга[3].
AutoDock поддерживается и разрабатывается лабораториями The Scripps Research Institute и Olson Laboratory[1].
Механизм работы
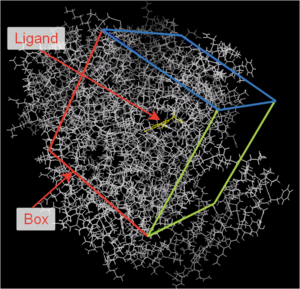
Докинг проводится в кубическую область внутри рецептора (docking box). С помощью AutoGrid для рецептора создается набор бинарных файлов — сеток потенциалов. Они описывают для каждого атома, входящего в docking box, потенциал его взаимодействия с пробным атомом определённого химического элемента. Набор этих элементов определяется химическим составом лигандов, с которыми требуется провести докинг. На каждый химический элемент создается по 1-2 файла. Для расчета потенциала используется скоринг функция, либо основанная на физических законах, либо эмпирическая, либо смешанная. Скоринг функции в разных версиях программы могут отличаться. Сетки потенциалов используются для расчета свободной энергии[4].
Лиганд, помимо совокупности атомов, связей и зарядов, внутри программы описывается набором чисел — положением в docking box, поворотом всех активных торсионных углов. Autodock перебирает все возможные комбинации этих чисел, чтобы в итоге найти оптимальное с точки зрения свободной энергии положение лиганда в docking box. Поэтому в качестве docking box обычно выбирают куб со стороной 10-30 ангстрем таким образом, чтобы он включал в себя активный центр рецептора[4].
Полный перебор всех возможных положений в активном центре и всех конформаций лиганда — очень время- и ресурсо-затратная задача. Для оптимизации процесса в Autodock применяются алгоритмы поиска глобального минимума: Монте-Карло, имитации отжига, генетические алгоритмы: LGA (Ламаркианский генетический алгоритм)[4].
Пример работы Autodock 4.2 на основе LGA[5]:
- Генерируется популяция лигандов (размер популяции задается пользователем). Каждый лиганд случайным образом расположен в docking boх, имеет случайную конформацию и описывается набором трансляционных, ориентационных и конформационных чисел.
- Для каждого элемента популяции рассчитывается свободная энергия. Она включает в себя энергию межмолекулярного взаимодействия рецептора с лигандом, и внутримолекулярную энергию лиганда.
- Стадия размножения. Для каждого элемента рассчитывается количество потомков по формуле: [math]\displaystyle{ n=(f{w}-f{i})/(f{w}-f{mean}) }[/math], где [math]\displaystyle{ n }[/math] – количество потомков, [math]\displaystyle{ f{i} }[/math] – свободная энергия элемента, [math]\displaystyle{ f{w} }[/math] – энергия худшего элемента (максимальная), [math]\displaystyle{ f{mean} }[/math] – средняя энергия. После этого каждый элемент лучше среднего получает пропорциональное [math]\displaystyle{ n }[/math] количество потомков, которые добавляются в популяцию.
- Стадия кроссинговера: случайным образом выбираются два элемента популяции и их характеристические числа рекомбинируют, то есть элементы обмениваются некоторыми из чисел. Процент рекомбинирующих пар задается пользователем.
- Стадия мутации: к характеристическим числам элемента прибавляется случайное число, соответствующее распределению Коши. Параметры распределения и процент мутаций задаются пользователем.
- Стадия вымирания: худшие элементы популяции удаляются. Количество выживших также задается пользователем.
- Стадия оптимизации. Каждый элемент популяции оптимизируется с помощью алгоритмов локального поиска, например, имитации отжига при низких температурах, что позволяет минимизировать свободную энергию в небольших пределах. Эта стадия отличает LGA от классических генетических алгоритмов.
- Шаги 2-7 повторяются определённое большое количество раз (пока лучшая свободная энергия не перестанет изменяться, либо при достижении лимита по времени или количеству итераций).
- Вся предыдущая последовательность шагов выполняется несколько раз (независимые запуски).
- Результаты множественных запусков кластеризуются по положению в docking box с отсечкой по RMSD 1 ангстрем.
- Для всех находок в кластере считается энергия взаимодействия с рецептором. Энергией кластера считается самая лучшая в этом кластере.
- Финальная энергия рассчитывается для лучших кластеров как разность между разностями энергий перехода рецептора с лигандом из несвязанного положения в связанное[6].
История версий
Autodock 1
Создан в 1990 году. Первая версия базируется на силовом поле Amber. Скоринг функция является суммой потенциала Леннарда-Джонса, электростатического потенциала и эмпирических функций энергии ковалентных связей, плоских и торсионных углов[7].
- [math]\displaystyle{ V=\sum_\text{bonds} k_b (l-l_0)^2 + \sum_\text{angles} k_a (\theta - \theta_0)^2 + \sum_\text{torsions} \sum_n \frac{1}{2} V_n [1+\cos(n \omega- \gamma)]+\sum_{j=1} ^{N-1} \sum_{i=j+1} ^N f_{ij}\biggl\{\epsilon_{ij}\biggl[\left(\frac{r_{0ij}}{r_{ij}} \right)^{12} - 2\left(\frac{r_{0ij}}{r_{ij}} \right)^{6} \biggr]+ \frac{q_iq_j}{4\pi \epsilon_0 r_{ij}}\biggr\} }[/math]
В реализации доступны алгоритмы Монте-Карло и имитации отжига[8].
Autodock 2.4
Появилось много улучшений, сопутствующих программ для параллельных запусков. Новый алгоритм для улучшения поиска, в случае лигандов с большим количеством степеней свободы, основан на принципе разделяй и властвуй. Улучшен алгоритм локальной оптимизации[9].
Autodock 3
Для оптимизации поиска впервые был применен комбинированный генетический алгоритм LGA[10]. Новая полуэмпирическая скоринг функция, калибрующаяся на 30 белок-лигандных комплексах, учитывает направленные водородные связи и энергию сольватации[5].
[math]\displaystyle{ V = W_{\text{vdw}}\sum\limits_{i,j}\left(\dfrac{A_{ij}}{r_{ij}^{12}} - \dfrac{B_{ij}}{r_{ij}^{6}}\right) +W_{\text{hbond}}\sum\limits_{i,j}E(t)\left(\dfrac{C_{ij}}{r_{ij}^{12}} - \dfrac{D_{ij}}{r_{ij}^{10}}\right) +W_{\text{elec}}\sum\limits_{i,j}\dfrac{q_iq_j}{e(r_{ij})r_{ij}}+ W_{\text{sol}}\sum\limits_{i,j}\left(S_iV_j - S_jV_i\right)e^{\left(-r_{ij}^2/2\sigma^2\right)} }[/math]
Autodock 4, 4.2
Добавлен учёт возможной подвижности боковых цепей. Это достигнуто за счет разделения белка в два файла. Одна часть считается статичной, другая подвижной. Со статической частью работают с помощью подсчета энергии AutoGrid, с подвижной работают теми же методами, что и с лигандом. Созданы новые типы атомов, например, галогены и основные ионы металлов. Улучшена скоринг функция. Калибровка на 250 структурах из PDBBind. Появился AutoDockTools — специальное программное обеспечение, для подготовки файлов к докингу[6].
Autodock 4.2.5
Добавлена более прозрачная опция контроля вывода программы, позволяющая делать небольшой вывод для скрининга, и выводить отчет для более глубокого анализа. Некоторые ошибки при исполнении программы, выводившие предупреждение в предыдущих версиях, теперь останавливают программу. Это сделано в ответ на нужды скрининга, при котором пользователь может не заметить появляющихся предупреждений. Учёт электростатических взаимодействий между не связанными атомами в лиганде теперь по умолчанию включен. Также включение/отключение возможно при помощи команды intelec on/off[4].
Autodock Vina
AutoDock Vina является новым поколением ПО, разработанным Molecular Graphics Lab. Показывает значительные улучшения средних показателей accuracy-метрики в предсказании сайтов связывания, так же заметно увеличение скорости работы в два раза, по сравнению с AutoDock 4.1. Принципиально новая скоринг функция, основанная на алгоритме X-Score[11], которая разработана с учётом развития мультипроцессорных систем. За счет различий в погрешностях и самих используемых функций скоринга, используемых в AutoDock 4 и AutoDock Vina, программы могут показывать различные результаты на одних данных[12].
AutoDockTools
Существует ассоциированный с Autodock графический интерфейс AutoDockTools (ADT), который помогает анализировать докинг и выбирать связи в лиганде, которые будут считаться подвижными. Ниже упомянуты некоторые из функций ADT[1]:
- Видеть и вращать молекулы в формате 3D;
- Добавлять молекулы водорода, как полярные, так и не полярные;
- Ассоциировать частичные заряды атомов с лигандами и макромолекулами;
- Обозначать подвижные связи в лиганде, с использованием графической версии AutoTors;
- Настраивать DPF, GPF файлы;
- Запускать AutoGrid и AutoDock;
- Выводить и визуализировать результат работы AutoDock.
Входные файлы
Файлы с расширением .pdb являются основным форматом хранения информации о конформации и строении молекул. Такие файлы получают из экспериментальных данных полученных рентгеновской кристаллографией или ЯМР-спектроскопией, либо методами предсказания структуры молекул. Начиная с версии AutoDock 4 процедура докинга требует два файла в формате .pdbqt; один для рецептора, другой для лиганда. Если в белке необходимо учитывать подвижность некоторых аминокислот, то создается третий файл, содержащий информацию об атомах в подвижных частях белка. Преобразование файлов .pdb в формат .pdbqt возможно с помощью программы AutoDock Tools[4].
Файлы pdbqt содержат следующую информацию[4]:
- Координаты атомов,
- Полярные водороды,
- Частичные заряды на атомах,
- Тип атомов, например, есть два разных типа для алифатического и ароматического углерода,
- Сведения о подвижных связях.
Интерпретация результатов
В результате работы Autodock для одного лиганда получается dlg файл. В нём записан подробный отчет о работе программы. Он содержит в себе результаты каждого конкретного запуска с конечным положением лиганда (структура лиганда записана в формате pdbqt), рассчитанной энергией, затраченным на расчет временем[4].
Также доступен результат кластеризации: для каждого кластера показана населенность (Num in cluster), лучшая энергия (Lowest binding energy), к какому конкретно запуску она относится (Run)[4]:
CLUSTERING HISTOGRAM
____________________
________________________________________________________________________________
| | | | |
Clus | Lowest | Run | Mean | Num | Histogram
-ter | Binding | | Binding | in |
Rank | Energy | | Energy | Clus| 5 10 15 20 25 30 35
_____|___________|_____|___________|_____|____:____|____:____|____:____|____:___
1 | -3.44 | 150 | -3.44 | 2 |##
2 | -3.42 | 63 | -3.41 | 42 |#x42
3 | -3.42 | 187 | -3.40 | 83 |#x83
4 | -3.38 | 115 | -3.36 | 33 |#x33
5 | -3.32 | 128 | -3.31 | 37 |#x37
6 | -3.28 | 122 | -3.27 | 3 |###
_____|___________|_____|___________|_____|______________________________________Собственно результатами являются энергия ΔG и положение лиганда в активном центре рецептора. Разница энергий лигандов в первом приближении показывает, насколько один лиганд лучше связывается рецептором, чем другой. Положение лиганда в активном центре позволяет предсказывать механизм связывания[13].
Области применения
AutoDock находит применение в следующих областях[1]:
- Дизайн лекарств,
- Исследования химических процессов.
Autodock широко применяется в научном сообществе как для молекулярного докинга, так и для виртуального скрининга больших библиотек соединений (например ZINC)[14].
Докинг используется также для поиска блокаторов ферментов патогенных организмов, в частности топоизомеразы I туберкулезной палочки[15].
Белок тирозинфосфатазa В Mycobacterium tuberculosis (Mtb) (MptpB) является важным фактором вирулентности для бактерии, который способствует выживанию бактерий в макрофагах. Отсутствие человеческого ортолога делает MptpB привлекательной мишенью для новых терапевтических средств для лечения туберкулеза. Ингибиторы MptpB могут быть эффективным средством для преодоления возникающей лекарственной устойчивости к туберкулезу. Использовав стратегию виртуального скрининга на основе структуры, авторы успешно идентифицировали лекарственный ингибитор MptpB на основе тиобарбитурата[16].
С помощью Autodock нашли ингибиторы ВИЧ протеазы[17]. В частности, ингибиторы аспарагиновой пептидазы ВИЧ (HIV IPs) являются хорошими кандидатами для повторного использования лекарственных средств.
Кроме того, докинг используется для поиска лигандов, взаимодействующих с транскрипционными факторами. Например, HNF-1a является транскрипционным фактором, который регулирует метаболизм глюкозы путем экспрессии в различных тканях. С помощью докинга in silico были найдены потенциальные мишени[18].
С помощью докинга были смоделированы взаимодействия двух регуляторов транскрипции, ExuR и UxuR, с субстратами и интермедиатами гликолиза, путей Эшвелла и Энтнера-Дудорова. Для UxuR были обнаружены два предпочтительных сайта связывания лиганда: один расположен в С-терминальном домене, а другой занимает междоменное пространство. Для ExuR в междоменной области был обнаружен только один предпочтительный сайт[19].
Autodock (в основном Vina) широко применяется в большом числе автоматических систем виртуального скрининга[20][21][22].
Сотрудничество с проектами
World Community Grid предлагает свою помощь в получении бесплатной вычислительной мощности для ускорения исследований, проводимых на основе AutoDock[1].
AutoDock запущен в работу на основе World Community Grid со следующими проектами:
- FightAIDS@Home от The Scripps Research Institute[23],
- Discover Dengue Drugs — Together от The University of Texas Medical Branch[24],
- Help Fight Childhood Cancer[25],
- Influenza Antiviral Drug Search[26],
- GO Fight Against Malaria[27].
Альтернативные программы
В 2016 году была проведена оценка различных программ на выборке из 2002 комплексов белка с лигандом. Оценивалась частота совпадений в найденных с помощью докинга позициях. Совпадением считались случаи, когда RMSD между найденным и нативным положением лиганда не превышало 2 Å[28][29].
Среди альтернативных академических программ выделяют LeDock, rDock, UCSF Dock, при этом первая программа показала наилучший результат (57,4% совпадения)[29].
Коммерческие альтернативные программы показали лучший результат (59,8% совпадения у GOLD), чем академические. Также в исследовании участвовали программы Surflex, FlexX, Glide, LigandFit, MOE-Dock, ICM_pro, MCDock, FRED.[28].
| Программа | Совпадение | Программа | Совпадение | |
|---|---|---|---|---|
| GOLD | 59,8 % | Autodock Vina | 49,0 % | |
| Glide (XP) | 57,8 % | AutoDock (PSO) | 47,3 % | |
| LeDock | 57,4 % | LigandFit | 46,1 % | |
| Glide (SP) | 53,8 % | MOE Dock | 45,6 % | |
| Surflex-Dock | 53,2 % | UCSD DOCK | 44,0 % | |
| rDock | 50,3% | AutoDock (LGA) | 37,4 % |
Примечания
- ↑ 1,0 1,1 1,2 1,3 1,4 1,5 Garrett M. Morris. AutoDock (англ.). autodock.scripps.edu. Дата обращения: 4 апреля 2017. Архивировано 13 января 2020 года.
- ↑ AutoDock Vina - molecular docking and virtual screening program. http://vina.scripps.edu.+Дата обращения: 3 мая 2019. Архивировано 8 января 2020 года.
- ↑ Sousa Sérgio Filipe, Fernandes Pedro Alexandrino, Ramos Maria João. Protein-ligand docking: Current status and future challenges (англ.) // Proteins: Structure, Function, and Bioinformatics. — 2006. — 21 July (vol. 65, no. 1). — P. 15—26. — ISSN 0887-3585. — doi:10.1002/prot.21082.
- ↑ 4,0 4,1 4,2 4,3 4,4 4,5 4,6 4,7 Garrett M. Morris, David S. Goodsell, Michael E. Pique, William “Lindy” Lindstrom, Ruth Huey, Stefano Forli, William E. Hart, Scott Halliday, Rik Belew and Arthur J. Olson. AutoDock4.2 User Guide. Дата обращения: 4 апреля 2017. Архивировано 18 мая 2017 года.
- ↑ 5,0 5,1 doi:10.1002/(SICI)1096-987X(19981115)19:14<1639::AID-JCC10>3.0.CO;2-B
Вы можете подставить цитату вручную или с помощью бота. - ↑ 6,0 6,1 Morris Garrett M., Huey Ruth, Lindstrom William, Sanner Michel F., Belew Richard K., Goodsell David S., Olson Arthur J. AutoDock4 and AutoDockTools4: Automated docking with selective receptor flexibility (англ.) // Journal of Computational Chemistry. — 2009. — December (vol. 30, no. 16). — P. 2785—2791. — ISSN 0192-8651. — doi:10.1002/jcc.21256.
- ↑ Goodford P. J. A computational procedure for determining energetically favorable binding sites on biologically important macromolecules. (англ.) // Journal Of Medicinal Chemistry. — 1985. — July (vol. 28, no. 7). — P. 849—857. — PMID 3892003.
- ↑ Goodsell David S., Olson Arthur J. Automated docking of substrates to proteins by simulated annealing (англ.) // Proteins: Structure, Function, and Genetics. — 1990. — Vol. 8, no. 3. — P. 195—202. — ISSN 0887-3585. — doi:10.1002/prot.340080302.
- ↑ Morris Garrett M., Goodsell David S., Huey Ruth, Olson Arthur J. Distributed automated docking of flexible ligands to proteins: Parallel applications of AutoDock 2.4 (англ.) // Journal of Computer-Aided Molecular Design. — 1996. — August (vol. 10, no. 4). — P. 293—304. — ISSN 0920-654X. — doi:10.1007/BF00124499.
- ↑ Fuhrmann Jan, Rurainski Alexander, Lenhof Hans-Peter, Neumann Dirk. A new Lamarckian genetic algorithm for flexible ligand-receptor docking (англ.) // Journal of Computational Chemistry. — 2010. — P. NA—NA. — ISSN 0192-8651. — doi:10.1002/jcc.21478.
- ↑ Wang R., Lai L., Wang S. Further development and validation of empirical scoring functions for structure-based binding affinity prediction. (англ.) // Journal Of Computer-aided Molecular Design. — 2002. — January (vol. 16, no. 1). — P. 11—26. — PMID 12197663.
- ↑ Trott Oleg, Olson Arthur J. AutoDock Vina: Improving the speed and accuracy of docking with a new scoring function, efficient optimization, and multithreading (англ.) // Journal of Computational Chemistry. — 2009. — P. NA—NA. — ISSN 0192-8651. — doi:10.1002/jcc.21334.
- ↑ Ma Xiaoli, Yan Jin, Xu Kailin, Guo Luiqi, Li Hui. Binding mechanism of trans-N-caffeoyltyramine and human serum albumin: Investigation by multi-spectroscopy and docking simulation (англ.) // Bioorganic Chemistry. — 2016. — June (vol. 66). — P. 102—110. — ISSN 0045-2068. — doi:10.1016/j.bioorg.2016.04.002.
- ↑ Anand R. Identification of Potential Antituberculosis Drugs Through Docking and Virtual Screening. (англ.) // Interdisciplinary Sciences, Computational Life Sciences. — 2018. — June (vol. 10, no. 2). — P. 419—429. — doi:10.1007/s12539-016-0175-6. — PMID 27147082.
- ↑ Sandhaus Shayna, Chapagain Prem P., Tse-Dinh Yuk-Ching. Discovery of novel bacterial topoisomerase I inhibitors by use of in silico docking and in vitro assays (англ.) // Scientific Reports. — 2018. — 23 January (vol. 8, no. 1). — ISSN 2045-2322. — doi:10.1038/s41598-018-19944-4.
- ↑ Zhang D., Lin Y., Chen X., Zhao W., Chen D., Gao M., Wang Q., Wang B., Huang H., Lu Y., Lu Y. Docking- and pharmacophore-based virtual screening for the identification of novel Mycobacterium tuberculosis protein tyrosine phosphatase B (MptpB) inhibitor with a thiobarbiturate scaffold. (англ.) // Bioorganic Chemistry. — 2019. — April (vol. 85). — P. 229—239. — doi:10.1016/j.bioorg.2018.12.038. — PMID 30641319.
- ↑ Schames Julie R., Henchman Richard H., Siegel Jay S., Sotriffer Christoph A., Ni Haihong, McCammon J. Andrew. Discovery of a Novel Binding Trench in HIV Integrase (англ.) // Journal of Medicinal Chemistry. — 2004. — April (vol. 47, no. 8). — P. 1879—1881. — ISSN 0022-2623. — doi:10.1021/jm0341913.
- ↑ Sridhar Gumpeny Ramachandra, Nageswara Rao Padmanabhuni Venkata, Kaladhar Dowluru SVGK, Devi Tatavarthi Uma, Kumar Sali Veeresh. In SilicoDocking of HNF-1a Receptor Ligands (англ.) // Advances in Bioinformatics. — 2012. — Vol. 2012. — P. 1—5. — ISSN 1687-8027. — doi:10.1155/2012/705435.
- ↑ Tutukina Maria N., Potapova Anna V., Vlasov Peter K., Purtov Yuri A., Ozoline Olga N. Structural modeling of the ExuR and UxuR transcription factors ofE. coli: search for the ligands affecting their regulatory properties (англ.) // Journal of Biomolecular Structure and Dynamics. — 2016. — 6 January (vol. 34, no. 10). — P. 2296—2304. — ISSN 0739-1102. — doi:10.1080/07391102.2015.1115779.
- ↑ Wójcikowski Maciej, Zielenkiewicz Piotr, Siedlecki Pawel. Open Drug Discovery Toolkit (ODDT): a new open-source player in the drug discovery field (англ.) // Journal of Cheminformatics. — 2015. — 22 June (vol. 7, no. 1). — ISSN 1758-2946. — doi:10.1186/s13321-015-0078-2.
- ↑ AutoDock | Raccoon2. autodock.scripps.edu. Дата обращения: 15 мая 2016. Архивировано 1 июня 2016 года.
- ↑ Sharma V., Pattanaik K. K., Jayprakash V., Basu A., Mishra N. A utility script for automating and integrating AutoDock and other associated programs for virtual screening. (англ.) // Bioinformation. — 2009. — 6 September (vol. 4, no. 2). — P. 84—86. — PMID 20198176.
- ↑ FightAIDS@Home. www.worldcommunitygrid.org. Дата обращения: 31 марта 2017. Архивировано 10 мая 2017 года.
- ↑ Discovering Dengue Drugs – Together. www.worldcommunitygrid.org. Дата обращения: 31 марта 2017. Архивировано 29 марта 2016 года.
- ↑ Help Fight Childhood Cancer. www.worldcommunitygrid.org. Дата обращения: 31 марта 2017. Архивировано 21 апреля 2017 года.
- ↑ Influenza Antiviral Drug Search. www.worldcommunitygrid.org. Дата обращения: 31 марта 2017. Архивировано 1 апреля 2017 года.
- ↑ Rarey. A fast flexible docking method using an incremental construction algorithm. Дата обращения: 19 апреля 2019. Архивировано 24 апреля 2019 года.
- ↑ 28,0 28,1 28,2 Wang Zhe, Sun Huiyong, Yao Xiaojun, Li Dan, Xu Lei, Li Youyong, Tian Sheng, Hou Tingjun. Comprehensive evaluation of ten docking programs on a diverse set of protein–ligand complexes: the prediction accuracy of sampling power and scoring power (англ.) // Physical Chemistry Chemical Physics. — 2016. — Vol. 18, no. 18. — P. 12964—12975. — ISSN 1463-9076. — doi:10.1039/c6cp01555g.
- ↑ 29,0 29,1 Pagadala Nataraj S., Syed Khajamohiddin, Tuszynski Jack. Software for molecular docking: a review (англ.) // Biophysical Reviews. — 2017. — 16 January (vol. 9, no. 2). — P. 91—102. — ISSN 1867-2450. — doi:10.1007/s12551-016-0247-1.
Ссылки
- Сайт разработчиков AutoDock. Дата обращения: 15 мая 2016. Архивировано 13 января 2020 года.