Спиральная катушка

Спиральная катушка — структурный мотив в белках, в котором 2-7[1] альфа-спиралей свернуты вместе, как нити веревки. (Димеры и тримеры — самые распространенные типы.) Многие белки типа спиральной катушки участвуют в важных биологических функциях, таких как регуляция экспрессии генов — например, факторы транскрипции. Яркими примерами являются онкопротеины c-Fos и c-jun, а также мышечный белок тропомиозин.
Открытие
Возможность формирования спиральных катушек для α-кератина изначально была несколько спорной. Линус Полинг и Фрэнсис Крик независимо друг от друга пришли к выводу, что это было возможно примерно в одно и то же время. Летом 1952 года Полинг посетил лабораторию в Англии, где работал Крик. Полинг и Крик встречались и говорили на разные темы; в какой-то момент Крик спросил, рассматривал ли Полинг «спиральные катушки» (Крик придумал этот термин), на что Полинг ответил, что так и есть. По возвращении в Соединенные Штаты Полинг возобновил исследования по этой теме. Он пришел к выводу, что спиральные катушки существуют, и в октябре отправил длинную рукопись в журнал Nature. Сын Полинга Питер Полинг работал в той же лаборатории, что и Крик, и рассказал ему об этом отчете. Крик считал, что Полинг украл его идею, и отправил более короткую заметку в Nature через несколько дней после получения рукописи Полинга. В конце концов, после некоторых разногласий и частых переписок, лаборатория Крика заявила, что эта идея была независимо достигнута обоими исследователями и что никакого интеллектуального воровства не произошло[2]. В своей заметке (которая была опубликована первой из-за её меньшей длины) Крик предложил спиральную катушку, а также математические методы определения их структуры[3]. Примечательно, что это произошло вскоре после того, как в 1951 году Линус Полинг и его коллеги предложили структуру альфа-спирали[4]. Эти исследования были опубликованы при отсутствии сведений о последовательности кератина. Первые последовательности кератина были определены Ханукоглу и Фуксом в 1982 г.[5][6]
На основе анализа предсказания последовательности и вторичной структуры идентифицированы домены спиральных кератинов[6]. Эти модели были подтверждены структурным анализом спиральных доменов кератинов[7].
Молекулярная структура
Спиральные катушки обычно содержат повторяющийся узор hxxhcxc из гидрофобных (h) и заряженных (c) аминокислотных остатков, называемый гептадным повтором[8]. Положения в гептадном повторе обычно обозначаются abcdefg, где a и d — гидрофобные положения, часто занятые изолейцином, лейцином или валином. Сворачивание последовательности с этим повторяющимся мотивом во вторичную альфа-спиральную структуру приводит к тому, что гидрофобные остатки будут представлены в виде «полосы», которая мягко наматывается вокруг спирали левозакрученным образом, образуя амфипатическую структуру. Наиболее благоприятный способ размещения двух таких спиралей в водонаполненной среде цитоплазмы — это наматывание гидрофобных цепей друг на друга, зажатых между гидрофильными аминокислотами. Таким образом, именно захоронение гидрофобных поверхностей обеспечивает термодинамическую движущую силу для олигомеризации. Упаковка на границе раздела спираль-спираль исключительно плотная, с почти полным ван-дер-ваальсовым контактом между боковыми цепями остатков a и d. Эта плотная упаковка была первоначально предсказана Фрэнсисом Криком в 1952 году[3] и называется «набивкой ручек в отверстия» .
α-спирали могут быть параллельными или антипараллельными и обычно имеют левозакрученную суперспираль (рис. 1). Несколько правозакрученных спиральных катушек также наблюдались в природе и в разработанных белках[9].
Биологические роли
Роль в ВИЧ-инфекции
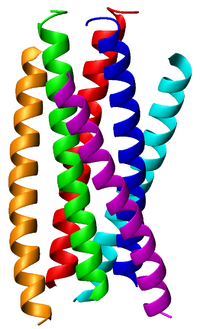
Проникновение вируса в CD4-положительные клетки начинается, когда три субъединицы гликопротеина 120 (gp120) связываются с рецептором CD4 и корецептором. Гликопротеин gp120 тесно связан с тримером gp41 посредством ван-дер-ваальсовых взаимодействий. При связывании gp120 с рецептором CD4 и корецептором ряд конформационных изменений в структуре приводит к диссоциации gp120 и экспонированию gp41 и в то же время к закреплению N-концевой слитой пептидной последовательности gp41 в клетка-хозяин. Подпружиненный механизм отвечает за то, чтобы мембраны вируса и клетки находились достаточно близко друг к другу, чтобы они могли слиться. Источник подпружиненного механизма лежит в экспонированном gp41, который содержит два последовательных гептадных повтора (HR1 и HR2), следующих за гибридным пептидом на N-конце белка. HR1 образует параллельную тримерную спиральную катушку, на которую наматывается область HR2, образуя структуру тримеров шпилек (или пучка из шести спиралей), тем самым облегчая слияние мембран за счет сближения мембран друг с другом. Затем вирус проникает в клетку и начинает репликацию. В последнее время ингибиторы получают из HR2, таких как Фузеон (DP178, Т-20) связываются с области HR1 gp41 на были разработаны. Однако пептиды, полученные из HR1, обладают небольшой эффективностью ингибирования вирусов из-за склонности этих пептидов к агрегации в растворе. Были разработаны химеры этих производных HR1 пептидов с лейциновыми застежками GCN4, которые, как было показано, более активны, чем Фузеон, но они ещё не вошли в клиническую практику.
Как метки олигомеризации
Из-за их специфического взаимодействия спиральные катушки могут использоваться в качестве «меток» для стабилизации или обеспечения определённого состояния олигомеризации[10]. Было обнаружено, что взаимодействие спиральной катушки управляет олигомеризацией субъединиц BBS2 и BBS7[11][12].
Дизайн
Общая проблема принятия решения о свернутой структуре белка при заданной аминокислотной последовательности (так называемая проблема укладки белка) не решена. Однако спиральная катушка представляет собой один из относительно небольшого числа мотивов складывания, для которых отношения между последовательностью и конечной складчатой структурой сравнительно хорошо поняты[13][14]. Harbury et al. выполнили знаковое исследование с использованием архетипической спиральной катушки, GCN4, в которой были установлены правила, которые регулируют то, как пептидная последовательность влияет на олигомерное состояние (то есть количество альфа-спиралей в окончательной сборке)[15][16]. Спиральная катушка GCN4 представляет собой 31-аминокислотную (что соответствует чуть более, чем четырём гептадам) параллельную, димерную (то есть состоящую из двух альфа-спиралей) спиральную катушку и имеет повторяющийся изолейцин (или I в однобуквенном коде) и лейцин (L) в положениях a и d, соответственно, и образует димерную спиральную катушку. Когда аминокислоты в положениях a и d были заменены с I на a и L на d на I на a и I на d, образовалась тримерная (три альфа-спирали) спиральная катушка. Кроме того, переключение положений L на a и с I на d привело к образованию тетрамерной (четыре альфа-спирали) спиральной катушки. Они представляют собой набор правил для определения олигомерных состояний спиральной катушки и позволяют ученым эффективно исследовать поведение олигомеризации. Другим аспектом сборки спиральной катушки, которая относительно хорошо изучена, по крайней мере, в случае димерных спиральных катушек, является то, что размещение полярного остатка (в частности, аспарагин, N) в противоположных положениях инициирует параллельную сборку спиральной катушки. Этот эффект обусловлен самокомплементарной водородной связью между этими остатками, которая не была бы удовлетворена, если бы N был спарен, например, с L на противоположной спирали[17].
Недавно Пикок, Пикрамену и его коллеги продемонстрировали, что спиральные катушки могут быть собраны самостоятельно, используя ионы лантаноида (III) в качестве матрицы, таким образом создавая новые агенты визуализации[18].
Примечания
- ↑ (Oct 2006) «A seven-helix coiled coil». Proceedings of the National Academy of Sciences of the United States of America 103 (42): 15457–62. doi:10.1073/pnas.0604871103. PMID 17030805. .
- ↑ Hager. Narrative 43, Coils Upon Coils. Linus Pauling and the Structure of Proteins. Oregon State University Special Collections and Archives Research Center. Дата обращения: 15 мая 2013. Архивировано 21 августа 2021 года.
- ↑ 3,0 3,1 (Nov 1952) «Is alpha-keratin a coiled coil?». Nature 170 (4334): 882–3. doi:10.1038/170882b0. PMID 13013241. .
- ↑ (Apr 1951) «The structure of proteins; two hydrogen-bonded helical configurations of the polypeptide chain». Proceedings of the National Academy of Sciences of the United States of America 37 (4): 205–11. doi:10.1073/pnas.37.4.205. PMID 14816373. .
- ↑ (Nov 1982) «The cDNA sequence of a human epidermal keratin: divergence of sequence but conservation of structure among intermediate filament proteins». Cell 31 (1): 243–52. doi:10.1016/0092-8674(82)90424-X. PMID 6186381.
- ↑ 6,0 6,1 (Jul 1983) «The cDNA sequence of a Type II cytoskeletal keratin reveals constant and variable structural domains among keratins». Cell 33 (3): 915–24. doi:10.1016/0092-8674(83)90034-X. PMID 6191871.
- ↑ (Jan 2014) «Proteopedia entry: coiled-coil structure of keratins». Biochemistry and Molecular Biology Education 42 (1): 93–4. doi:10.1002/bmb.20746. PMID 24265184.
- ↑ (Feb 2004) «Coiled coil domains: stability, specificity, and biological implications». ChemBioChem 5 (2): 170–6. doi:10.1002/cbic.200300781. PMID 14760737.
- ↑ (Nov 1998) «High-resolution protein design with backbone freedom». Science 282 (5393): 1462–7. doi:10.1126/science.282.5393.1462. PMID 9822371.
- ↑ (June 2014) «Your personalized protein structure: Andrei N. Lupas fused to GCN4 adaptors». Journal of Structural Biology 186 (3): 380–5. doi:10.1016/j.jsb.2014.01.013. PMID 24486584.
- ↑ Chou (3 September 2019). «The Molecular Architecture of Native BBSome Obtained by an Integrated Structural Approach». Structure 27 (9): 1384–1394. doi:10.1016/j.str.2019.06.006. PMID 31303482.
- ↑ Ludlam (17 September 2019). «Molecular architecture of the Bardet-Biedl syndrome protein 2-7-9 subcomplex.». The Journal of Biological Chemistry 294 (44): 16385–16399. doi:10.1074/jbc.RA119.010150. PMID 31530639.
- ↑ (Jan 2008) «Peptide and protein building blocks for synthetic biology: from programming biomolecules to self-organized biomolecular systems». ACS Chemical Biology 3 (1): 38–50. doi:10.1021/cb700249v. PMID 18205291.
- ↑ (May 2011) «Complex networks govern coiled-coil oligomerization--predicting and profiling by means of a machine learning approach». Molecular & Cellular Proteomics 10 (5): M110.004994. doi:10.1074/mcp.M110.004994. PMID 21311038.
- ↑ (Nov 1993) «A switch between two-, three-, and four-stranded coiled coils in GCN4 leucine zipper mutants». Science 262 (5138): 1401–7. doi:10.1126/science.8248779. PMID 8248779. .
- ↑ (Sep 1994) «Crystal structure of an isoleucine-zipper trimer». Nature 371 (6492): 80–3. doi:10.1038/371080a0. PMID 8072533. .
- ↑ Woolfson (2005). «The design of coiled-coil structures and assemblies». Adv. Protein. Chem. 70 (4): 79–112. doi:10.1016/S0065-3233(05)70004-8. PMID 15837514.
- ↑ (Jan 2014) «De novo design of Ln(III) coiled coils for imaging applications». Journal of the American Chemical Society 136 (4): 1166–9. doi:10.1021/ja408741h. PMID 24405157.
Дальнейшее чтение
- (1953) «The Packing of α-Helices: Simple Coiled-Coils». Acta Crystallogr 6 (8): 689–697. doi:10.1107/S0365110X53001964.
- (1976) «Geometrical criteria for formation of coiled-coil structures of polypeptide chains». Macromolecules 9 (3): 395–407. doi:10.1021/ma60051a004. PMID 940353. .
- (Nov 1993) «A switch between two-, three-, and four-stranded coiled coils in GCN4 leucine zipper mutants». Science 262 (5138): 1401–7. doi:10.1126/science.8248779. PMID 8248779. .
- (Jun 1996) «An engineered allosteric switch in leucine-zipper oligomerization». Nature Structural Biology 3 (6): 510–5. doi:10.1038/nsb0696-510. PMID 8646536.
- (Nov 1998) «High-resolution protein design with backbone freedom». Science 282 (5393): 1462–7. doi:10.1126/science.282.5393.1462. PMID 9822371.
- (Oct 2002) «Coiled-coils: stability, specificity, and drug delivery potential». Advanced Drug Delivery Reviews 54 (8): 1113–29. doi:10.1016/S0169-409X(02)00058-3. PMID 12384310.
- (May 2002) «Improving coiled-coil stability by optimizing ionic interactions». Journal of Molecular Biology 318 (3): 901–10. doi:10.1016/S0022-2836(02)00114-6. PMID 12054832.
- (Aug 2003) «Long coiled-coil proteins and membrane traffic». Biochimica et Biophysica Acta (BBA) - Molecular Cell Research 1641 (2–3): 71–85. doi:10.1016/S0167-4889(03)00088-0. PMID 12914949.
- (Feb 2004) «Coiled coil domains: stability, specificity, and biological implications». ChemBioChem 5 (2): 170–6. doi:10.1002/cbic.200300781. PMID 14760737.
Ссылки
Программное обеспечение, связанное со Спиральными катушками
Прогнозирование, обнаружение и визуализация
- Paircoil2/Paircoil
- bCIPA оценивает значения Tm для пар витых катушек
- Экран библиотеки bCIPA Проверяет библиотеку последовательностей по одной определённой цели и оценивает значения Tm для всех пар спиральных катушек.
- Экран bCIPA Interactome Отображает все взаимодействия между выбранными определёнными последовательностями и оценивает значения Tm для всех пар спиральных катушек.
- STRAP содержит алгоритм для предсказания витков на основе AA-последовательностей.
- PrOCoil предсказывает олигомеризацию белков спиральной катушки и визуализирует вклад каждой отдельной аминокислоты в общую олигомерную тенденцию.
- DrawCoil создает диаграммы спиральных колес для спиральных катушек любого состояния и ориентации олигомеризации.
Базы данных
- Spiricoil использует аннотацию белкового домена для прогнозирования наличия спиральной спирали и состояния олигорфизма для всех полностью секвенированных организмов.
- CC+ — это реляционная база данных спиральных катушек, найденных в PDB.
- Аннотации белковых доменов SUPERFAMILY для всех полностью секвенированных организмов на основе тщательно подобранного класса спиральной спирали SCOP
