Cytoscape
| Cytoscape | |
|---|---|
| Тип | Обработка изображений |
| Автор | Institute of Systems Biology |
Cytoscape ([ˌsaɪtə(u)'skeɪp], сайтоскейп, в русском биоинформатическом сленге — цитоскейп) — биоинформатическая платформа с открытым исходным кодом, предназначенная для визуализации сетей молекулярных взаимодействий и биологических путей с возможностью использования дополнительных данных, таких как функциональная аннотация, информация об уровне экспрессии генов и прочих. Несмотря на то, что исходно Cytoscape был разработан для биологических исследований, сейчас он широко используется для решения различных задач по анализу сетей и их визуализации[1].
История
Cytoscape был разработан в Институте системной биологии в Сиэтле в 2002 году. Первая версия программы, Cytoscape 0.8[2], была выпущена в июле 2002 года, последующие релизы 0.9 и 1.0 последовали в ноябре 2002 и марте 2003 соответственно. Серия 2.0 обновлялась с 2004 до 2012 года, после чего в 2013 году было положено начало серии 3.xx. Последнее большое обновление программы вышло в 2014 году с выходом Cytoscape v3.3, в котором были реорганизованы основные модели внутренних данных и значительно улучшена поддержка сторонних приложений и плагинов[3].
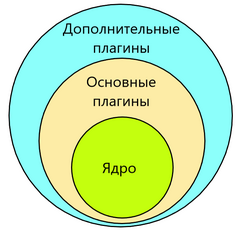
До версии 3.3 Cytoscape состоял из Ядра и дополнительных Плагинов. Начиная с версии 3.3, некоторые функции Ядра были выделены в основные Плагины. Ядро — это код, который организует, отображает, считывает и записывает сети, но не содержит биологических функций. Основные Плагины — это предустановленные модули, выполняющие функции, отличные от Ядра, но необходимые для использования Cytoscape. В отличие от предыдущих версий Cytoscape, функционал основных Плагинов можно обновлять, не выпуская нового большого обновления Cytoscape[3]. Также существуют дополнительные плагины (доступны в App Store), позволяющие выполнять определённые задачи[4]. С версии 3.6.1 Cytoscape автоматически устанавливает актуальную версию Java для работы[3].
Сейчас поддержку программы осуществляет Международный консорциум разработчиков программ с открытым кодом. На сегодняшний день последней версией Cytoscape является версия 3.8. Она содержит улучшения производительности и возможностей сетевого рендеринга и улучшение интеграции сервиса NDEx. Новая версия получила возможности использования для воспроизводимого высокопроизводительного анализа, мультимасштабных сетей, сетей белок-белковых взаимодействий, доступного анализа омики[англ.][3].
Одним из долгосрочных планов является тесная интеграция Cytoscape и NDEx, чтобы пользователи естественным образом загружали и хранили свои сети, используя сервис NDEx[3].
В настоящее время Cytoscape экспортирует сеть в виде файла, который можно использовать в качестве дополнительного материала для статьи в журнале. Онлайн-средство для просмотра статей издателя журнала может свободно отображать сеть в интерактивном режиме просмотра, который позволяет пользователю увеличивать масштаб сети и перемещать узлы[3].
Применение
Несмотря на то, что среда моделирования Cytoscape разработана для биологических задачах, данная программа может применяться во многих других областях[1].
В биологии
|
Конкретные примеры из статей 2019 года:
|
В социологии
|
Комплексный анализ сетей |
Возможности Cytoscape
Базовые возможности
Основная часть Cytoscape[1] представляет собой сетевой граф с молекулярными видами в качестве узлов (вершин) и межмолекулярных взаимодействий в качестве связей (ребер) между узлами. Cytoscape Core предоставляет базовую функциональность для интеграции произвольных данных на графе, визуального представления графа и интегрированных данных, инструментов выделения и фильтрации и интерфейса для внешних методов, реализованных в виде подключаемых модулей[8].
Визуализация сетей
В Cytoscape[1] возможны разнообразные способы визуального представления сетей (графов): циклический, в виде дерева, force-directed[9] и пр. Также пользователь может по-своему организовывать анализируемую сеть. Наложенные на сеть уровни экспрессии и значения p-value могут быть отображены в виде цвета вершин или ребер, толщины или цвета краевых линий и пр. Пользователь может воспользоваться готовыми схемами визуализации и настроить их самостоятельно[10].

Интеграция данных
Данные интегрируются с графической моделью с помощью атрибутов (Attributes). Это пары (имя, значение), которые сопоставляют имена узлов или ребер определенным значениям данных. Значения атрибута могут принимать любой тип (например, текстовые строки, дискретные или непрерывные числа, URL-адреса или списки) и либо загружаются из хранилища данных, либо генерируются динамически в сеансе. Графические браузеры позволяют пользователю просматривать все атрибуты выбранных узлов и ребер[11].
Передача аннотаций
В то время как атрибут является предикатом узла или ребра, Annotation представляет иерархическую классификацию (то есть, формально, ориентированный граф без циклов) описаний групп узлов или ребер. Аннотации обычно соответствуют существующему хранилищу знаний, которое является большим, сложным и относительно статическим. Cytoscape объединяет аннотации с другими сетевыми типами данных путем передачи желаемых уровней аннотации на атрибуты узлов или ребер. Используя контроллер аннотаций, можно иметь множество уровней аннотации активными и отображаемыми одновременно, каждый как отдельный атрибут на требуемых узлах или ребрах[12].
Визуализация графа
Одним из наиболее фундаментальных инструментов для интерпретации данных молекулярного взаимодействия является визуализация узлов и ребер в виде двумерной сети. Cytoscape поддерживает множество алгоритмов автоматизированной компоновки сети, включая иерархическую и круговую компоновки[13].
Визуализация атрибутов
Визуализация таких атрибутов как экспрессия генов и p-value. Cytoscape поддерживает широкий спектр визуальных свойств, таких как цвет, форма и размер узла, цвет и толщина границы узла, цвет края, толщина и стиль. Визуализация атрибута происходит с использованием таблицы поиска или интерполяции в зависимости от того, является атрибут дискретным или непрерывным[12].
Поиск и фильтрация частей графа
Чтобы уменьшить сложность сети с большим молекулярным взаимодействием, необходимо отображать подмножества узлов и ребер выборочно. Узлы и ребра могут быть выбраны в соответствии с широким рядом критериев, включая выбор по имени, по списку имен или на основе атрибута. Более сложные запросы выбора сети поддерживаются с помощью набора инструментов фильтрации, который включает фильтр минимальных соседних узлов, выбирающий узлы с минимальным количеством соседей на заданном расстоянии в сети; фильтр локального расстояния, который выбирает узлы на заданном расстоянии от группы предварительно выбранных узлов; комбинированный фильтр, который выбирает узлы произвольно и/или с помощью комбинаций других фильтров, и другие. Cytoscape позволяет производить поиск вершин или ребер по их названиям[13][14].
Выделение вершин или ребер
Пользователь может выделить подсеть вершин и/или ребер, обладающих какими-либо свойствами. Например, пользователь может выбрать все вершины, имеющие степень больше установленного порога, имеющие определенную функциональную аннотацию, или все вершины, обозначающие гены, чей уровень экспрессии сильно изменился хотя бы в одном эксперименте в соответствие с p-value, загруженными вместе с данными об уровнях экспрессии. Пользователь может создать новую сеть, выделив часть предыдущей[13].
Поиск модулей и кластеров
При исследовании сетей взаимодействия генов Cytoscape делает возможным поиск отдельных участков, которые состоят из генов с высокой экспрессионной активностью. Более того, в любом изучаемом объекте можно осуществлять поиск участков с высокой связностью элементов, или кластеров[13].

Поддержка многих форматов
Cytoscape поддерживает много стандартных форматов, передающих взаимодействия молекул и их аннотацию: SIF (Simple Interaction Format), GML, XGMML, BioPAX, PSI-MI, GraphML, KGML (KEGG XML), SBML, OBO и Gene Association. Текстовые файлы с разделителями, а также формат Microsoft Excel поддерживаются программой. Пользователь может импортировать файлы, содержащие данные об уровнях экспрессии генов и GO аннотацию, загружать и сохранять произвольные атрибуты на вершинах и ребрах сети (графа). Например, к вершинам, обозначающим белки, может быть приписана их функция, а к ребрам, обозначающим взаимодействия между белками, достоверность этих взаимодействий, эту информацию можно получить из базы данных STRING[13].
Функциональная совместимость
В силу того, что Cytoscape поддерживает широкий спектр форматов, его легко можно совмещать с другими программами. Например, если пользователь работал с сетями в программах igraph или Bioconductor, Cytoscape может загрузить файлы выдачи этих программ, провести анализ и сохранить результаты, например, в формате PSI-MI, который дальше может быть использован для обработки другими биоинформатическими программами или скриптами[13].
Связь со внешними базами данных
Cytoscape способен напрямую подключаться к сторонним базам данных, загружать сети и аннотацию. Примеры используемых баз данных: Pathway Commons, IntAct, BioMart, NCBI Entrez Gene[13].
Сохранение сессии
Текущее состояние работы может быть сохранено в виде файла сессии, который включает в себя все настройки, анализируемые сети, их визуализацию, стили, состояние рабочего окна, плагинов и пр. Файл сессии имеет расширение Cytoscape Session (.cys)[13].
Сохранение изображений
Cytoscape позволяет сохранять изображения в высоком качестве. Поддерживает следующие форматы: PDF, PS, SVG, PNG, JPEG и BMP[13].
Просмотр
Просмотр сетей в Cytoscape облегчен возможностью увеличения и уменьшения изображений, их прокрутки, ручного редактирования. Чтобы упростить работу с огромными сетями (например, содержащими более 100 000 вершин или ребер), имеется окно «вида с птичьего полета»[13].
Менеджер приложений и Cytoscape App Store
К данной программе доступны приложения для исследования сетей и молекулярных профилей. Cytoscape создан на платформе Java, поэтому можно создавать дополнительные приложения для импорта данных, их анализа и визуализации. Множество приложений доступны на Cytoscape App Store. Большинство приложений можно устанавливать с помощью менеджера App Manager или напрямую из App Store[15][13].
Поддержка других языков
Cytoscape использует 2-битовый стандарт кодирования символов. Можно использовать любой язык в файлах с данными. Многие функции и приложения Cytoscape поддерживают несколько языков, включая русский и восточно-азиатские[13].
Плагины Cytoscape
Подключаемые модули представляют собой мощные средства расширения возможностей для реализации новых алгоритмов, дополнительного сетевого анализа и биологической семантики. Подключаемые модули получают доступ к сетевой модели Core и могут также управлять отображением сети. Хотя Cytoscape Core — это открытое программное обеспечение, плагины — это отдельное программное обеспечение, которое может быть защищено любой лицензией в зависимости от авторов плагинов[16][13].
WikiPathways
WikiPathways — это база данных биологических путей, поддерживаемая научным сообществом. Каждый путь в базе данных снабжен идентификаторами, которые можно использовать для вычислений и визуализации данных. Плагин WikiPathways для Cytoscape доступен в App Store[17].
Legend Creator
Плагин для создания легенд в Cytoscape. Он добавляет панель управления, которая может сканировать сеть и таблицу стилей для создания легенды для каждой характеристики. Его основной функцией является добавление цветового градиента для определения количественного значения атрибутов[18][19].
GNC
GNC — новый плагин Cytoscape для оценки биологической когерентности генных сетей путем сравнения со стандартной. GNC плагин использует GNC алгоритм для оценки биологической когерентности генных сетей. Плагин был интегрирован в Cytoscape для увеличения доступности алгоритма для пользователей. Эта интеграция позволила пользователю анализировать не только глобальную биологическую согласованность сети, но и биологическую согласованность на уровне взаимоотношений генов. Это позволяет пользователю использовать Cytoscape для дальнейшего анализа и визуализации сетей[20].
ReNE
ReNE — плагин Cytoscape 3.x разработанный для автоматического обогащения стандартной регуляторной сети, созданной на основе генов с более подробными транскрипционными, посттранскрипционными и трансляционными данными. В результате получается расширенная сеть, которая точнее моделирует фактические биологические регуляторные механизмы. ReNE может автоматически импортировать сетевой макет из Reactome или KEGG или работать с пользовательскими путями, описанными с использованием стандартного формата данных OWL / XML, который принимает процедура импорта Cytoscape. Кроме того, ReNE позволяет исследователям объединять несколько путей, поступающих из разных источников. Получающаяся в результате расширенная сеть по-прежнему является полностью функциональной сетью Cytoscape, где каждый регуляторный элемент (фактор транскрипции, miRNA, ген, белок) и регуляторный механизм (повышающая регуляция / понижающая регуляция) четко визуально идентифицируются, что позволяет лучше визуально понять их роль и влияние на поведение сети. Усовершенствованная сеть, созданная ReNE, экспортируется в различные форматы для дальнейшего анализа через сторонние приложения. ReNE расширяет сеть, интегрируя данные только из общедоступных источников, без каких-либо выводов или предсказаний[21].
NOA
NOA — это плагин Cytoscape, используемый для анализа онтологии сетей. Реализованный алгоритм NOA основан на обогащении сетей, путем расширения аннотаций Gene Ontology до ссылок на сети или грани графа. Этот плагин облегчает анализ одной или нескольких сетей Cytoscape в соответствии с заданными пользователем параметрами. Плагин представляет результаты в виде таблиц, а также формирует тепловые карты и составляет обзор сетей из Cytoscape[22].
ClusterMaker
ClusterMaker — плагин Cytoscape реализующий несколько алгоритмов кластеризации и визуализации, которые можно использовать независимо или в комбинации для анализа и визуализации наборов биологических данных, а также для подтверждения или создания гипотез о биологической функции. Плагин предоставляет результаты в виде сети, дендрограммы и тепловой карты[23].
CytoCluster
CytoCluster — плагин Cytoscape для кластерного анализа биологических сетей. CytoCluster объединяет шесть алгоритмов кластеризации. А именно: HC-PIN (алгоритм иерархической кластеризации сетей взаимодействий белков), OH-PIN (идентификация перекрывающихся и иерархических модулей сетей взаимодействий белков), IPCA (алгоритм идентификации комплексных белков), ClusterONE (кластеризация с расширением перекрывающихся соседей), функция DCU (обнаружение комплексов на основе модели неопределенного графа), IPC-MCE (идентификация белковых комплексов на основе комплекса максимального расширения) и BinGO (онтология генов биологических сетей). Пользователь может выбрать любой из перечисленных алгоритмов кластеризации в соответствии со своими требованиями. Основная функция этих шести алгоритмов кластеризации заключена в обнаружении белковых комплексов или функциональных модулей. Более того, BinGO можно использовать для определения того, какие категории Gene Ontology (GO) статистически представлены несколько раз в наборе генов или подграфе биологической сети[24].
StringApp
STRING является одним из самых популярных источников белковых сетей, однако её веб-интерфейс в основном предназначен для проверки небольших сетей и соответствующих доказательств. Программное обеспечение Cytoscape намного лучше подходит для работы с большими сетями и предлагает большую гибкость в плане сетевого анализа, импорта и визуализации дополнительных данных. В связи с этим, был создан плагин stringApp, объединяющий Cytoscape STRING. И тем самым упрощает импорт сетей STRING в Cytoscape, сохраняет внешний вид и многие функции STRING, а также интегрирует данные из связанных баз данных[25].
CyClust3D
CyClust3D — плагин для кластеризации мотивов сетей интегрированных в сети. Традиционные алгоритмы кластеризации графов не могут обнаружить плотные топологические структуры или функциональные модули, в которые собираются мотивы сетей интегрированные в молекулярные сети. Плагин CyClust3D позволяет обнаруживать такие модули с помощью кластеризации составных трехузловых сетевых мотивов с использованием алгоритма трехмерной спектральной кластеризации[26].
PiNGO
PiNGO — плагин Cytoscape для поиска генов кандидатов в биологические сети. PiNGO представляет собой инструмент для скрининга биологических сетей на наличие генов-кандидатов, то есть генов, которые, как предсказывают, будут вовлечены в интересующий биологический процесс. Пользователь может сузить поиск до генов с конкретными известными функциями или исключить гены, принадлежащие к определенным функциональным классам. PiNGO обеспечивает поддержку широкого спектра организмов и схем классификации генной онтологии, и его можно легко настроить для других организмов и функциональных классификаций[27].
Примечания
- ↑ Перейти обратно: 1,00 1,01 1,02 1,03 1,04 1,05 1,06 1,07 1,08 1,09 1,10 1,11 1,12 1,13 1,14 1,15 1,16 1,17 Cytoscape: An Open Source Platform for Complex Network Analysis and Visualization (англ.). Дата обращения: 4 мая 2019. Архивировано 25 апреля 2019 года.
- ↑ Cytoscape App Store - gexf-app (англ.). Дата обращения: 4 мая 2019. Архивировано 25 апреля 2017 года.
- ↑ Перейти обратно: 3,0 3,1 3,2 3,3 3,4 3,5 Cytoscape Product Roadmap (англ.). cytoscape.org. Дата обращения: 29 апреля 2019. Архивировано 30 апреля 2019 года.
- ↑ Cytoscape App Store (англ.). apps.cytoscape.org. Дата обращения: 30 апреля 2019. Архивировано 30 апреля 2019 года.
- ↑ Wu Fan, Gao Feng, He Siyi, Xiao Yingbin. Identification of hub genes in chronically hypoxic myocardium using bioinformatics analysis (англ.) // Molecular Medicine Reports. — 2019. — 1 March. — ISSN 1791-2997. — doi:10.3892/mmr.2019.10001.
- ↑ Cooke Mariana, Casado-Medrano Victoria, Ann Jihyae, Lee Jeewoo, Blumberg Peter M., Abba Martin C., Kazanietz Marcelo G. Differential Regulation of Gene Expression in Lung Cancer Cells by Diacyglycerol-Lactones and a Phorbol Ester Via Selective Activation of Protein Kinase C Isozymes (англ.) // Scientific Reports. — 2019. — 15 April (vol. 9, no. 1). — ISSN 2045-2322. — doi:10.1038/s41598-019-42581-4.
- ↑ Lu Yang, Wang Xinmin, Dong Hongchang, Wang Xiaofang, Yang Pu, Han Ling, Wang Yingzi, Zheng Zhihong, Zhang Wanjiang, Zhang Le. Bioinformatics analysis of microRNA expression between patients with and without latent tuberculosis infections (англ.) // Experimental and Therapeutic Medicine. — 2019. — 20 March. — ISSN 1792-0981. — doi:10.3892/etm.2019.7424.
- ↑ Tutorial (англ.). Open Tutorials. Дата обращения: 3 мая 2019. (недоступная ссылка)
- ↑ Tutorial (англ.). Open Tutorials. Дата обращения: 4 мая 2019. (недоступная ссылка)
- ↑ Tutorial (англ.). OpenTutorials. Дата обращения: 4 мая 2019. (недоступная ссылка)
- ↑ Tutorial (англ.). OpenTutorials. Дата обращения: 4 мая 2019. (недоступная ссылка)
- ↑ Перейти обратно: 12,0 12,1 Tutorial (англ.). OpenTutorials. Дата обращения: 4 мая 2019. (недоступная ссылка)
- ↑ Перейти обратно: 13,00 13,01 13,02 13,03 13,04 13,05 13,06 13,07 13,08 13,09 13,10 13,11 13,12 What is Cytoscape? (англ.). cytoscape.org. Дата обращения: 3 мая 2019. Архивировано 27 ноября 2018 года.
- ↑ Tutorial (англ.). Open Tutorials. Дата обращения: 3 мая 2019. (недоступная ссылка)
- ↑ Saito Rintaro, Smoot Michael E, Ono Keiichiro, Ruscheinski Johannes, Wang Peng-Liang, Lotia Samad, Pico Alexander R, Bader Gary D, Ideker Trey. A travel guide to Cytoscape plugins (англ.) // Nature Methods. — 2012. — November (vol. 9, no. 11). — P. 1069—1076. — ISSN 1548-7091. — doi:10.1038/nmeth.2212.
- ↑ Tutorial (англ.). OpenTutorials. Дата обращения: 3 мая 2019. (недоступная ссылка)
- ↑ WikiPathways App (англ.). cytoscape.org. Дата обращения: 30 апреля 2019. Архивировано 28 мая 2019 года.
- ↑ Legend Creator Tutorial (англ.). cytoscape.org. Дата обращения: 30 апреля 2019. Архивировано 30 апреля 2019 года.
- ↑ Cytoscape App Store - Legend Creator (англ.). apps.cytoscape.org. Дата обращения: 30 апреля 2019. Архивировано 30 апреля 2019 года.
- ↑ Díaz-Montaña Juan J., Gómez-Vela Francisco, Díaz-Díaz Norberto. GNC–app: A new Cytoscape app to rate gene networks biological coherence using gene–gene indirect relationships (англ.) // Biosystems. — 2018. — April (vol. 166). — P. 61—65. — ISSN 0303-2647. — doi:10.1016/j.biosystems.2018.01.007.
- ↑ Politano Gianfranco, Benso Alfredo, Savino Alessandro, Di Carlo Stefano. ReNE: A Cytoscape Plugin for Regulatory Network Enhancement (англ.) // PLoS ONE. — 2014. — 26 December (vol. 9, no. 12). — P. e115585. — ISSN 1932-6203. — doi:10.1371/journal.pone.0115585.
- ↑ Zhang Chao, Wang Jiguang, Hanspers Kristina, Xu Dong, Chen Luonan, Pico Alexander R. NOA: a cytoscape plugin for network ontology analysis (англ.) // Bioinformatics. — 2013. — 7 June (vol. 29, no. 16). — P. 2066—2067. — ISSN 1460-2059. — doi:10.1093/bioinformatics/btt334.
- ↑ Morris John H, Apeltsin Leonard, Newman Aaron M, Baumbach Jan, Wittkop Tobias, Su Gang, Bader Gary D, Ferrin Thomas E. clusterMaker: a multi-algorithm clustering plugin for Cytoscape (англ.) // BMC Bioinformatics. — 2011. — 9 November (vol. 12, no. 1). — ISSN 1471-2105. — doi:10.1186/1471-2105-12-436.
- ↑ Li Min, Li Dongyan, Tang Yu, Wu Fangxiang, Wang Jianxin. CytoCluster: A Cytoscape Plugin for Cluster Analysis and Visualization of Biological Networks (англ.) // International Journal of Molecular Sciences. — 2017. — 31 August (vol. 18, no. 9). — P. 1880. — ISSN 1422-0067. — doi:10.3390/ijms18091880.
- ↑ Doncheva Nadezhda T., Morris John H., Gorodkin Jan, Jensen Lars J. Cytoscape StringApp: Network Analysis and Visualization of Proteomics Data (англ.) // Journal of Proteome Research. — 2018. — 19 November (vol. 18, no. 2). — P. 623—632. — ISSN 1535-3893. — doi:10.1021/acs.jproteome.8b00702.
- ↑ Audenaert Pieter, Van Parys Thomas, Brondel Florian, Pickavet Mario, Demeester Piet, Van de Peer Yves, Michoel Tom. CyClus3D: a Cytoscape plugin for clustering network motifs in integrated networks (англ.) // Bioinformatics. — 2011. — 8 April (vol. 27, no. 11). — P. 1587—1588. — ISSN 1460-2059. — doi:10.1093/bioinformatics/btr182.
- ↑ Smoot M., Ono K., Ideker T., Maere S. PiNGO: a Cytoscape plugin to find candidate genes in biological networks (англ.) // Bioinformatics. — 2011. — 28 January (vol. 27, no. 7). — P. 1030—1031. — ISSN 1367-4803. — doi:10.1093/bioinformatics/btr045.
Ссылки
- cytoscape.org — официальный сайт Cytoscape
- Shannon P., Markiel A., Ozier O., Baliga N. S., Wang J. T., Ramage D., Amin N., Schwikowski B., Ideker T. Cytoscape: a software environment for integrated models of biomolecular interaction networks. (англ.) // Genome Research. — 2003. — November (vol. 13, no. 11). — P. 2498—2504. — doi:10.1101/gr.1239303. — PMID 14597658.