Секвенирование экзома

Секвени́рование экзо́ма (англ. Exome sequencing) — секвенирование всех белок-кодирующих генов в геноме (то есть экзома). Под секвенированием экзома подразумеваются две операции: во-первых, отбор экзонов. В зависимости от организма экзоны покрывают 1—2 % генома[1]. У человека их насчитывается около 180 000, примерно 1 % от всего генома, или приблизительно 30 миллионов пар оснований (п. о.). Во-вторых, секвенирование экзонов с использованием любой платформы высокопроизводительного секвенирования ДНК и анализ полученных результатов[2].
Секвенирование экзома позволяет обнаружить генетические изменения, приводящие к изменению белковых последовательностей, которые могут в свою очередь приводить к возникновению заболеваний, таких как атеросклероз, болезнь Альцгеймера и других. Основное преимущество экзомного секвенирования заключается в возможности проводить массовый скрининг генов и обнаруживать мутации, ассоциированные с заболеваниями, при этом данная процедура оказывается проще и дешевле, чем полногеномное секвенирование[1].
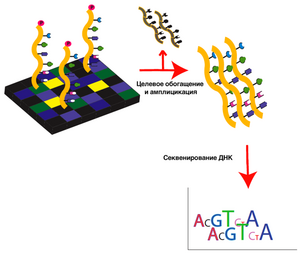
Методология
Секвенирование экзома включает в себя четыре этапа: выделение ДНК из предоставленного материала, отбор интересующей фракции ДНК (обогащение образцов), секвенирование отобранного материала и анализ полученных результатов[3].
Выделение ДНК
Первый этап заключается в подготовке высококачественных препаратов геномной ДНК из предоставленных образцов путем отделения ДНК от белков, липидов и т.д. Стандартный метод выделения ДНК— экстракция смесью фенол-хлороформ[4].
Стратегии обогащения образцов
Стратегии обогащения образцов позволяют селективно отбирать нужные геномные участки, то есть экзоны, из образцов ДНК до этапа секвенирования. С момента описания первого оригинального метода в 2005 году разработано несколько стратегий обогащения образцов, подходящих для целей экзомного секвенирования[5]. Выбор конкретного метода зависит от размеров интересующих участков, потребности в покрытии при секвенировании, имеющегося в наличии оборудования и других причин[6].
Полимеразная цепная реакция
Полимеразная цепная реакция (ПЦР) широко применяется для амплификации требуемых фрагментов ДНК уже более 20 лет[7]. Обычно в ПЦР используют только 2 праймера, однако разработаны методы мультиплексной ПЦР, которая использует несколько праймеров и позволяет одновременно амплифицировать несколько ДНК-мишеней в ходе одного процесса. Подходы, использующие ПЦР, очень эффективны, но не позволяют работать с участками генома длиной в несколько миллионов п.о. из-за высокой цены и низкого качества получающихся образцов[1].
Метод молекулярной инверсии

Метод молекулярной инверсии — это техника, которая позволяет получить образцы ДНК, обогащенные амплифицированными инвертированными участками целевых последовательностей. Отбор нужных последовательностей происходит за счет замыкания интересующего участка в кольцо. Праймер здесь представляет собой одноцепочечный ДНК-олигонуклеотид, в центральной части которого содержится универсальная последовательность с сайтами рестрикции, а концы комплементарны двум участкам геномной ДНК, между которыми находится интересующая последовательность. Образцы, не вступившие в реакцию, остаются линейными и удаляются экзонуклеазами[5][8]. Метод может быть полезен для работы с небольшим числом мишеней в большом количестве образцов. Главный недостаток — единообразие получаемых образцов, а также высокая цена при необходимости покрыть большой набор участков[7].
Гибридизационное обогащение
Для гибридизационного обогащения образцов экзомными участками создаются специальные микрочипы, содержащие закрепленные на подложке одноцепочечные олигонуклеотиды (зонды) с последовательностями из генома, способными покрыть интересующие участки. Геномная ДНК разрезается на фрагменты. Концы фрагментов делают тупыми с помощью рестриктаз, добавляют адаптеры с универсальными праймерами. После гибридизации фрагментов с зондами на микрочипах негибридизованные фрагменты отмываются с подложки, а оставшиеся затем амплифицируются с помощью ПЦР[5]. Ограничения метода связаны с дороговизной аппаратуры, количеством зондов, которые можно разместить на матрице, и необходимостью достаточно больших количеств ДНК для анализа[1].
Обогащение в растворе
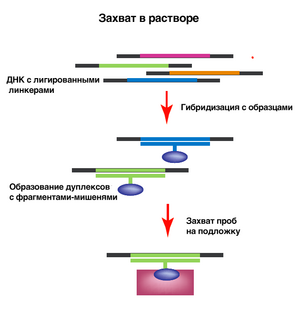
В растворе синтезируется набор зондов, которые фиксируются на стрептавидиновых шариках. Шарики помещаются в раствор с фрагментированной геномной ДНК, где происходит селективная гибридизация зондов с нужными геномными участками, после чего шарики с интересующими фрагментами осаждают и отмывают. Затем оставшиеся участки секвенируют. Этот метод был разработан для усовершенствования метода гибридизационного обогащения: он позволяет создать избыток зондов к целевым участкам по сравнению с необходимым количеством образца. Оптимальный размер целевого участка ДНК — около 3,5 миллиона п.о., так при последующем секвенировании получается хорошее покрытие[7].
Платформы, используемые для обогащения экзома
Основными поставщиками платформ для обогащения экзома являются NimbleGen, Agilent и Illumina[1].
| NimbleGen’s SeqCap EZ Exome Library | Agilent’s Sure Select Human All Exon Kit | Illumina’s TruSeq Exome Enrichment Kit | Illumina’s Nextera Rapid Capture Exome Kit | |
|---|---|---|---|---|
| Длина зондов | 55 — 105[9] | 114 — 126[9] | 95 | 95 |
| Рекомендованное количество ДНК пробы | 3 мкг[10] | 3 мкг[10] | 500 нг[10] | 50 нг[10] |
| Тип нуклеиновой кислоты зонда | ДНК | РНК | ДНК | ДНК |
| Стратегия покрытия интересующего фрагмента зондами | Перекрывающиеся зонды[9] | Чаще строго последовательные зонды, чем перекрывающиеся | Гэпы между последовательностями зондов (зонды находятся на некотором расстоянии друг от друга по последовательности фрагмента) | Гэпы между последовательностями зондов |
| Метод фрагментации | Ультразвук | Ультразвук | Ультразвук | Транспозаза |
| Размер целевого фрагмента (для человека) | 64 | 50 | 62 | 62 |
| Прочтения, остающиеся после фильтрации | 66 % | 71,7 % | 54,8 %[11] | 40,1% |
| Основные сильные стороны | Высокая чувствительность и специфичность. Наиболее равномерное покрытие в трудных регионах[9][12][13]. | Хорошее покрытие инделов[9][13][11]. Высокая скорость выравнивания. Меньше повторных прочтений, чем у других платформ[13]. | Хорошее покрытие нетранслируемых областей и микроРНК[9] | Хорошее покрытие нетранслируемых областей и микроРНК |
| Основные слабые стороны | Больше повторных чтений, чем у Agilent. Меньшая скорость выравнивания. | Меньше качественных чтений, чем у NimbleGen[12] | Высокий уровень нецелевого обогащения[9] | Высокий уровень нецелевого обогащения. Смещение покрытия для областей с высоким GC-составом, снижающим однородность. |
| Использование не только для человеческих последовательностей | Да | Да | Нет | Нет |
В настоящее время, в дополнение к наборам, нацеленным лишь на человека, NimbleGen предлагает наборы для экзомов кукурузы, ячменя, пшеницы, сои, мыши и свиньи, а Agilent — для экзомов мыши, крупного рогатого скота и рыбок данио-рерио. Оба поставщика также предлагают возможность разрабатывать индивидуальные комплекты для других видов. Наборы для нечеловеческих видов используют протоколы и зонды, аналогичные человеческим наборам поставщиков. Оба производителя предлагают гибкий процесс проектирования, который позволяет вносить изменения для улучшения покрытия для конкретных регионов и целей[1].
Секвенирование
Существует несколько технологий секвенирования, включая классический метод секвенирования по Сэнгеру. Методы секвенирования нового поколения используют платформы Illumina, SOLiD и Ion-Torrent. Все эти методы могут использоваться в том числе и для секвенирования экзома[14].
Анализ результатов
Первичные данные секвенирования представляют собой огромный набор небольших последовательностей (чтений), длина и качество которых зависят от технических характеристик секвенатора и способа приготовления образцов. Качество чтений можно контролировать, например, при помощи программного пакета FastQС[15]. Полученные чтения фильтруются: отрезаются концевые участки, которые часто имеют большое число ошибок, удаляются адаптерные последовательности (например, с помощью Trimmomatic[16] или sickle[17]); затем корректируются ошибки (например, с помощью программ Bloocoo[18] и Lighter[19]). Отфильтрованные чтения картируются на геном, где собираются в последовательности, соответствующие экзонам. В настоящий момент существует множество программ, которые осуществляют каждый этап подготовки данных секвенирования и их анализа, большинство из них требует больших вычислительных мощностей, так как объём получаемых данных очень велик[20].
Применение экзомного секвенирования
Используя экзомное секвенирование, в ходе исследований с фиксированными затратами мы можем секвенировать последовательности с существенно большей глубиной покрытия по сравнению с покрытием, получаемым методами полногеномного секвенирования. Благодаря этому экзомное секвенирование чаще используется при решении задач, требующих надежного определения однонуклеотидных полиморфизмов[21].
Клиническая диагностика
29 сентября 2011 года компания Ambry Genetics стала первой сертифицированной компанией, предлагающей секвенирование экзома и диагностику заболеваний на его основе[22]. В компании утверждают, что результаты экзомного секвенирования позволят сотрудникам диагностировать заболевания, при которых традиционные диагностические подходы неприменимы[23].
Идентификация мутаций, вызывающих заболевания, может внести существенный вклад в диагностические и терапевтические подходы, поможет прогнозировать развитие заболевания и позволит тестировать родственников, находящихся в зоне риска[2][24][25][26][27][28]. Есть несколько факторов, на основании которых экзомному секвенированию отдается предпочтение перед моногенным анализом: возможность идентифицировать мутации в генах, не подвергшихся тестированию ввиду нетипичного клинического проявления[28] и идентификация клинических случаев, при которых мутации в разных генах вызывают различные проявления у одного и того же пациента[24]. Кроме того, метод позволяет диагностировать заболевания на ранних этапах и у молодых пациентов до проявления всего спектра характерных симптомов; он также используется для пренатальной диагностики[1] В некоторых случаях пренатальное секвенирование экзома позволяет выявить генетические заболевания, в то время как стандартные методы (кариотипирование и использование микрочипов) оказываются неэффективны[29].
Авторы важнейшей рецензированной публикации об экзомном секвенировании подчеркивают полезность этого метода для клинической практики. Авторы, применившие экзомное секвенирование для идентификации мутации, вызывающей синдром Барттера и конгенитальную хлоридную диарею, заявляют: «Мы видим будущее, в котором подобная информация станет частью повседневной клинической оценки пациентов с подозрениями на генетические заболевания с неясным диагнозом… Мы предвидим, что полноэкзомное секвенирование внесет огромный вклад в понимание того, какие гены и какими путями участвуют в развитии редких и частых человеческих болезней, а также в клиническую практику»[25].
Картирование редких полиморфизмов при комплексных расстройствах и менделевских болезнях
Текущие крупные международные исследования направлены на определение в геноме частых полиморфизмов, которые легче всего идентифицировать современными методами. Однако из-за отрицательного отбора полиморфизмы, вызывающие крайне тяжелые заболевания, в частности, менделевские болезни, встречаются с существенно меньшей аллельной частотой и могут остаться невыявленными в ходе поиска генов-кандидатов при использовании современных стандартных методов генотипирования*, при этом чаще всего они расположены в границах экзома. Так как при комплексных расстройствах с риском заболевания связано большое количество генов, для обнаружения их необходимы исследования очень большой выборки, поэтому, с точки зрения издержек, полногеномное секвенирование не является оптимальным. К тому же, полиморфизмы в кодирующих областях изучаются очень подробно, и их функциональное значение проще определить[30] Успешная модель идентификации менделевских генов включает определение возникающих de novo полиморфизмов при секвенировании генов двух родителей и потомка[31].
Использование в сельском хозяйстве
Геномы растений могут быть чрезвычайно сложными, повторяющимися и часто полиплоидными; в результате некоторые из наиболее экономически важных культур не удается исследовать с использованием полногеномного секвенирования. Разработан набор для обогащения экзома пшеницы на основе накопленных данных транскриптома[32], с использованием которого были проведены исследования нежелательной внутрикультурной генетической гетерогенности экзома, влияющей на фенотип растения, в частности, скорость роста, способность жить в различных условиях и другие важные для селекции признаки. Подобные же наборы были использованы при исследовании риса Oryza sativa[33] и сои Glycine max[34]. Также можно идентифицировать генетические маркеры, отвечающие за особую устойчивость растительных культур к определенным патогенам[35].
В ряде случаев секвенирование экзома может быть использовано как альтернатива более дорогому секвенированию полного генома, например, при исследовании генетических вариаций внутри и между популяциями[36].
Сравнение с генотипированием с использованием микрочипов
Методы микрочипирования требуют зондов для гибридизации с известной последовательностью, поэтому они ограничены требованиями к разработке зондов и не позволяют выявить некоторые генетические изменения. Технологии высокопроизводительного секвенирования, используемые для секвенирования экзома, позволяют узнать последовательности гораздо большего числа локусов одновременно и определить неизвестные до сих пор источники многих болезней[37], то есть позволяют обойти ограничения генотипирующих чипов и классического секвенирования[38].
Секвенирование экзома — процедура более дорогая, но по мере уменьшения финансовых затрат и увеличения производительности методов секвенирования этот метод все шире используется в практике для диагностики редких генетических заболеваний[39].
Ограничения
Некоторые болезни могут быть связаны с мутациями в некодирующих областях или структурными перестройками, которые секвенирование экзома не позволит выявить[2]. Но из-за дороговизны полногеномного секвенирования на нынешнем этапе развития науки и технологий экзомное секвенирование представляется оптимальным методом для клинической диагностики редких наследственных заболеваний, не выявляемых микрочипами[25].
Статистический анализ больших объёмов данных в ходе секвенирования экзома — отдельная трудоемкая задача. Есть несколько подходов для улучшения качества экзомных данных[2]:
- сравнение полиморфизмов, определённых с помощью секвенирования и микрочипирования;
- сравнение кодирующих полиморфизмов с данными полногеномного секвенирования пациентов с таким же заболеванием;
- сравнение кодирующих ОНП с гаплотипами, отсеквенированными по Сэнгеру.
Для некоторых биологических видов качество сборки генома и его аннотация значительно хуже, чем для человека (или секвенированного генома нет вовсе). Это существенно ограничивает применение секвенирования экзома к другим организмам, поскольку осложняет обогащение образцов ДНК и картирование результатов секвенирования на геном[1].
Примечания
- ↑ 1,0 1,1 1,2 1,3 1,4 1,5 1,6 1,7 1,8 Warr Amanda, Robert Christelle, Hume David, Archibald Alan, Deeb Nader, Watson Mick. Exome Sequencing: Current and Future Perspectives (англ.) // G3: Genes. — 2015. — 2 July (vol. 5, no. 8). — P. 1543—1550. — ISSN 2160-1836. — doi:10.1534/g3.115.018564.
- ↑ 2,0 2,1 2,2 2,3 Ng Sarah B., Turner Emily H., Robertson Peggy D., Flygare Steven D., Bigham Abigail W., Lee Choli, Shaffer Tristan, Wong Michelle, Bhattacharjee Arindam, Eichler Evan E., Bamshad Michael, Nickerson Deborah A., Shendure Jay. Targeted capture and massively parallel sequencing of 12 human exomes (англ.) // Nature. — 2009. — 16 August (vol. 461, no. 7261). — P. 272—276. — ISSN 0028-0836. — doi:10.1038/nature08250.
- ↑ Whole Exome Sequencing | Detect exonic variants. www.illumina.com. Дата обращения: 5 мая 2019. Архивировано 3 мая 2019 года.
- ↑ Psifidi Androniki, Dovas Chrysostomos I., Bramis Georgios, Lazou Thomai, Russel Claire L., Arsenos Georgios, Banos Georgios. Comparison of Eleven Methods for Genomic DNA Extraction Suitable for Large-Scale Whole-Genome Genotyping and Long-Term DNA Banking Using Blood Samples (англ.) // PLOS ONE. — 2015. — 30 January (vol. 10, no. 1). — P. e0115960. — ISSN 1932-6203. — doi:10.1371/journal.pone.0115960.
- ↑ 5,0 5,1 5,2 Turner Emily H., Ng Sarah B., Nickerson Deborah A., Shendure Jay. Methods for Genomic Partitioning (англ.) // Annual Review of Genomics and Human Genetics. — 2009. — September (vol. 10, no. 1). — P. 263—284. — ISSN 1527-8204. — doi:10.1146/annurev-genom-082908-150112.
- ↑ Mamanova Lira, Coffey Alison J, Scott Carol E, Kozarewa Iwanka, Turner Emily H, Kumar Akash, Howard Eleanor, Shendure Jay, Turner Daniel J. Target-enrichment strategies for next-generation sequencing (англ.) // Nature Methods. — 2010. — February (vol. 7, no. 2). — P. 111—118. — ISSN 1548-7091. — doi:10.1038/nmeth.1419.
- ↑ 7,0 7,1 7,2 Kahvejian Avak, Quackenbush John, Thompson John F. What would you do if you could sequence everything? (англ.) // Nature Biotechnology. — 2008. — October (vol. 26, no. 10). — P. 1125—1133. — ISSN 1087-0156. — doi:10.1038/nbt1494.
- ↑ Mertes F., ElSharawy A., Sauer S., van Helvoort J. M. L. M., van der Zaag P. J., Franke A., Nilsson M., Lehrach H., Brookes A. J. Targeted enrichment of genomic DNA regions for next-generation sequencing (англ.) // Briefings in Functional Genomics. — 2011. — 1 November (vol. 10, no. 6). — P. 374—386. — ISSN 2041-2649. — doi:10.1093/bfgp/elr033.
- ↑ 9,0 9,1 9,2 9,3 9,4 9,5 9,6 Clark Michael J, Chen Rui, Lam Hugo Y K, Karczewski Konrad J, Chen Rong, Euskirchen Ghia, Butte Atul J, Snyder Michael. Performance comparison of exome DNA sequencing technologies (англ.) // Nature Biotechnology. — 2011. — 25 September (vol. 29, no. 10). — P. 908—914. — ISSN 1087-0156. — doi:10.1038/nbt.1975.
- ↑ 10,0 10,1 10,2 10,3 Seaby Eleanor G., Pengelly Reuben J., Ennis Sarah. Exome sequencing explained: a practical guide to its clinical application (англ.) // Briefings in Functional Genomics. — 2015. — 9 December (vol. 15, no. 5). — P. 374—384. — ISSN 2041-2649. — doi:10.1093/bfgp/elv054.
- ↑ 11,0 11,1 Chilamakuri Chandra Sekhar, Lorenz Susanne, Madoui Mohammed-Amin, Vodák Daniel, Sun Jinchang, Hovig Eivind, Myklebost Ola, Meza-Zepeda Leonardo A. Performance comparison of four exome capture systems for deep sequencing (англ.) // BMC Genomics. — 2014. — Vol. 15, no. 1. — P. 449. — ISSN 1471-2164. — doi:10.1186/1471-2164-15-449.
- ↑ 12,0 12,1 Sulonen Anna-Maija, Ellonen Pekka, Almusa Henrikki, Lepistö Maija, Eldfors Samuli, Hannula Sari, Miettinen Timo, Tyynismaa Henna, Salo Perttu, Heckman Caroline, Joensuu Heikki, Raivio Taneli, Suomalainen Anu, Saarela Janna. Comparison of solution-based exome capture methods for next generation sequencing (англ.) // Genome Biology. — 2011. — Vol. 12, no. 9. — P. R94. — ISSN 1465-6906. — doi:10.1186/gb-2011-12-9-r94.
- ↑ 13,0 13,1 13,2 Bodi K., Perera A. G., Adams P. S., Bintzler D., Dewar K., Grove D. S., Kieleczawa J., Lyons R. H., Neubert T. A., Noll A. C., Singh S., Steen R., Zianni M. Comparison of Commercially Available Target Enrichment Methods for Next-Generation Sequencing (англ.) // Journal of Biomolecular Techniques : JBT. — 2013. — July (vol. 24, no. 2). — P. 73—86. — ISSN 1524-0215. — doi:10.7171/jbt.13-2402-002.
- ↑ Quail Michael, Smith Miriam E, Coupland Paul, Otto Thomas D, Harris Simon R, Connor Thomas R, Bertoni Anna, Swerdlow Harold P, Gu Yong. A tale of three next generation sequencing platforms: comparison of Ion torrent, pacific biosciences and illumina MiSeq sequencers (англ.) // BMC Genomics. — 2012. — Vol. 13, no. 1. — P. 341. — ISSN 1471-2164. — doi:10.1186/1471-2164-13-341.
- ↑ Пакет FastQC (англ.). Babraham Bioinformatics. Дата обращения: 30 апреля 2015. Архивировано 2 марта 2019 года.
- ↑ Пакет Trimmomatic (англ.). USADELLAB.org. Дата обращения: 30 апреля 2015. Архивировано 22 апреля 2015 года.
- ↑ Пакет sicke (англ.). GitHub. Дата обращения: 30 апреля 2015. Архивировано 27 апреля 2015 года.
- ↑ Программа Bloocoo (англ.). Inria. Дата обращения: 30 апреля 2015. Архивировано 4 марта 2016 года.
- ↑ Программа Lighter (англ.). GitHub. Дата обращения: 30 апреля 2015. Архивировано 11 июля 2017 года.
- ↑ Mardis Elaine R. The challenges of big data (англ.) // Disease Models & Mechanisms. — 2016. — 1 May (vol. 9, no. 5). — P. 483—485. — ISSN 1754-8403. — doi:10.1242/dmm.025585.
- ↑ Pengelly Reuben J, Gibson Jane, Andreoletti Gaia, Collins Andrew, Mattocks Christopher J, Ennis Sarah. A SNP profiling panel for sample tracking in whole-exome sequencing studies (англ.) // Genome Medicine. — 2013. — Vol. 5, no. 9. — P. 89. — ISSN 1756-994X. — doi:10.1186/gm492.
- ↑ Ambry Genetics. Ambry Genetics First to Offer Exome Sequencing Service for Clinical Diagnostics. (англ.). www.prnewswire.com. Дата обращения: 5 мая 2019. Архивировано 16 апреля 2017 года.
- ↑ Volk Amber, Conboy Erin, Wical Beverly, Patterson Marc, Kirmani Salman. Whole-Exome Sequencing in the Clinic: Lessons from Six Consecutive Cases from the Clinician's Perspective (англ.) // Molecular Syndromology. — 2015. — 3 February (vol. 6, no. 1). — P. 23—31. — ISSN 1661-8769. — doi:10.1159/000371598.
- ↑ 24,0 24,1 Ng Sarah B, Buckingham Kati J, Lee Choli, Bigham Abigail W, Tabor Holly K, Dent Karin M, Huff Chad D, Shannon Paul T, Jabs Ethylin Wang, Nickerson Deborah A, Shendure Jay, Bamshad Michael J. Exome sequencing identifies the cause of a mendelian disorder (англ.) // Nature Genetics. — 2009. — 13 November (vol. 42, no. 1). — P. 30—35. — ISSN 1061-4036. — doi:10.1038/ng.499.
- ↑ 25,0 25,1 25,2 Choi Murim, Scholl Ute I., Ji Weizhen, Liu Tiewen, Tikhonova Irina R., Zumbo Paul, Nayir Ahmet, Bakkaloğlu Ayșin, Özen Seza, Sanjad Sami, Nelson-Williams Carol, Farhi Anita, Mane Shrikant, Lifton Richard P. Genetic diagnosis by whole exome capture and massively parallel DNA sequencing (англ.) // Proceedings of the National Academy of Sciences. — 2009. — 27 October (vol. 106, no. 45). — P. 19096—19101. — ISSN 0027-8424. — doi:10.1073/pnas.0910672106.
- ↑ Bilgüvar Kaya, Öztürk Ali Kemal, Louvi Angeliki, Kwan Kenneth Y., Choi Murim, Tatlı Burak, Yalnızoğlu Dilek, Tüysüz Beyhan, Çağlayan Ahmet Okay, Gökben Sarenur, Kaymakçalan Hande, Barak Tanyeri, Bakırcıoğlu Mehmet, Yasuno Katsuhito, Ho Winson, Sanders Stephan, Zhu Ying, Yılmaz Sanem, Dinçer Alp, Johnson Michele H., Bronen Richard A., Koçer Naci, Per Hüseyin, Mane Shrikant, Pamir Mehmet Necmettin, Yalçınkaya Cengiz, Kumandaş Sefer, Topçu Meral, Özmen Meral, Šestan Nenad, Lifton Richard P., State Matthew W., Günel Murat. Whole-exome sequencing identifies recessive WDR62 mutations in severe brain malformations (англ.) // Nature. — 2010. — 22 August (vol. 467, no. 7312). — P. 207—210. — ISSN 0028-0836. — doi:10.1038/nature09327.
- ↑ Worthey Elizabeth A, Mayer Alan N, Syverson Grant D, Helbling Daniel, Bonacci Benedetta B, Decker Brennan, Serpe Jaime M, Dasu Trivikram, Tschannen Michael R, Veith Regan L, Basehore Monica J, Broeckel Ulrich, Tomita-Mitchell Aoy, Arca Marjorie J, Casper James T, Margolis David A, Bick David P, Hessner Martin J, Routes John M, Verbsky James W, Jacob Howard J, Dimmock David P. Making a definitive diagnosis: Successful clinical application of whole exome sequencing in a child with intractable inflammatory bowel disease (англ.) // Genetics in Medicine. — 2010. — 17 December (vol. 13, no. 3). — P. 255—262. — ISSN 1098-3600. — doi:10.1097/GIM.0b013e3182088158.
- ↑ 28,0 28,1 Raffan Eleanor, Hurst Liam A., Turki Saeed Al, Carpenter Gillian, Scott Carol, Daly Allan, Coffey Alison, Bhaskar Sanjeev, Howard Eleanor, Khan Naz, Kingston Helen, Palotie Aarno, Savage David B., O'Driscoll Mark, Smith Claire, O'Rahilly Stephen, Barroso Inês, Semple Robert K. Early Diagnosis of Werner's Syndrome Using Exome-Wide Sequencing in a Single, Atypical Patient (англ.) // Frontiers in Endocrinology. — 2011. — Vol. 2. — ISSN 1664-2392. — doi:10.3389/fendo.2011.00008.
- ↑ Best Sunayna, Wou Karen, Vora Neeta, Van der Veyver Ignatia B., Wapner Ronald, Chitty Lyn S. Promises, pitfalls and practicalities of prenatal whole exome sequencing (англ.) // Prenatal Diagnosis. — 2017. — 25 July (vol. 38, no. 1). — P. 10—19. — ISSN 0197-3851. — doi:10.1002/pd.5102.
- ↑ Nickerson Sarah, Marquis-Nicholson Renate, Claxton Karen, Ashton Fern, Leong Ivone, Prosser Debra, Love Jennifer, George Alice, Taylor Graham, Wilson Callum, Gardner R., Love Donald. SNP Analysis and Whole Exome Sequencing: Their Application in the Analysis of a Consanguineous Pedigree Segregating Ataxia (англ.) // Microarrays. — 2015. — 23 October (vol. 4, no. 4). — P. 490—502. — ISSN 2076-3905. — doi:10.3390/microarrays4040490.
- ↑ Lee Hane, Deignan Joshua L., Dorrani Naghmeh, Strom Samuel P., Kantarci Sibel, Quintero-Rivera Fabiola, Das Kingshuk, Toy Traci, Harry Bret, Yourshaw Michael, Fox Michelle, Fogel Brent L., Martinez-Agosto Julian A., Wong Derek A., Chang Vivian Y., Shieh Perry B., Palmer Christina G. S., Dipple Katrina M., Grody Wayne W., Vilain Eric, Nelson Stanley F. Clinical Exome Sequencing for Genetic Identification of Rare Mendelian Disorders (англ.) // JAMA. — 2014. — 12 November (vol. 312, no. 18). — P. 1880. — ISSN 0098-7484. — doi:10.1001/jama.2014.14604.
- ↑ Winfield Mark O., Wilkinson Paul A., Allen Alexandra M., Barker Gary L. A., Coghill Jane A., Burridge Amanda, Hall Anthony, Brenchley Rachael C., D’Amore Rosalinda, Hall Neil, Bevan Michael W., Richmond Todd, Gerhardt Daniel J., Jeddeloh Jeffrey A., Edwards Keith J. Targeted re-sequencing of the allohexaploid wheat exome (англ.) // Plant Biotechnology Journal. — 2012. — 18 June (vol. 10, no. 6). — P. 733—742. — ISSN 1467-7644. — doi:10.1111/j.1467-7652.2012.00713.x.
- ↑ Henry I. M., Nagalakshmi U., Lieberman M. C., Ngo K. J., Krasileva K. V., Vasquez-Gross H., Akhunova A., Akhunov E., Dubcovsky J., Tai T. H., Comai L. Efficient Genome-Wide Detection and Cataloging of EMS-Induced Mutations Using Exome Capture and Next-Generation Sequencing (англ.) // The Plant Cell. — 2014. — 1 April (vol. 26, no. 4). — P. 1382—1397. — ISSN 1040-4651. — doi:10.1105/tpc.113.121590.
- ↑ Bolon Y.-T., Haun W. J., Xu W. W., Grant D., Stacey M. G., Nelson R. T., Gerhardt D. J., Jeddeloh J. A., Stacey G., Muehlbauer G. J., Orf J. H., Naeve S. L., Stupar R. M., Vance C. P. Phenotypic and Genomic Analyses of a Fast Neutron Mutant Population Resource in Soybean (англ.) // PLANT PHYSIOLOGY. — 2011. — 14 February (vol. 156, no. 1). — P. 240—253. — ISSN 0032-0889. — doi:10.1104/pp.110.170811.
- ↑ Wendler Neele, Mascher Martin, Nöh Christiane, Himmelbach Axel, Scholz Uwe, Ruge-Wehling Brigitte, Stein Nils. Unlocking the secondary gene-pool of barley with next-generation sequencing (англ.) // Plant Biotechnology Journal. — 2014. — 6 July (vol. 12, no. 8). — P. 1122—1131. — ISSN 1467-7644. — doi:10.1111/pbi.12219.
- ↑ Song Shen, Yao Na, Yang Min, Liu Xuexue, Dong Kunzhe, Zhao Qianjun, Pu Yabin, He Xiaohong, Guan Weijun, Yang Ning, Ma Yuehui, Jiang Lin. Exome sequencing reveals genetic differentiation due to high-altitude adaptation in the Tibetan cashmere goat (Capra hircus) (англ.) // BMC Genomics. — 2016. — 18 February (vol. 17, no. 1). — ISSN 1471-2164. — doi:10.1186/s12864-016-2449-0.
- ↑ Biesecker Leslie G. Exome sequencing makes medical genomics a reality (англ.) // Nature Genetics. — 2010. — January (vol. 42, no. 1). — P. 13—14. — ISSN 1061-4036. — doi:10.1038/ng0110-13.
- ↑ Berberich Amanda J., Ho Rosettia, Hegele Robert A. Whole genome sequencing in the clinic: empowerment or too much information? (англ.) // Canadian Medical Association Journal. — 2018. — 2 February (vol. 190, no. 5). — P. E124—E125. — ISSN 0820-3946. — doi:10.1503/cmaj.180076.
- ↑ DNA Sequencing Costs: Data | NHGRI. www.genome.gov. Дата обращения: 6 мая 2019. Архивировано 6 мая 2019 года.