ДНК-маркер

ДНК-ма́ркеры (ДНК-маркёры), или молекулярно-генетические маркеры — полиморфный признак, выявляемый методами молекулярной биологии на уровне нуклеотидной последовательности ДНК для определенного гена или для любого другого участка хромосомы при сравнении генотипов различных особей, пород, сортов, линий.
За последние годы накопилось много данных об эффективности использования молекулярно-генетических маркеров на уровне как белков, так и ДНК, РНК, для решения многих задач генетики, селекции, сохранения биоразнообразия, изучения механизмов эволюции, картирования хромосом, а также для семеноводства и племенного дела.
Наиболее широко используемые молекулярно-генетические маркеры условно можно подразделить на следующие типы — маркеры участков структурных генов, кодирующих аминокислотные последовательности белков (электрофоретические варианты белков), маркеры некодирующих участков структурных генов и маркеры различных последовательностей ДНК, отношение которых к структурным генам, как правило, неизвестно — распределение коротких повторов по геному (RAPD — случайно амплифицируемая полиморфная ДНК; ISSR — инвертированные повторы; AFLP — полиморфизм в сайтах рестрикции) и микросателлитные локусы (тандемные повторы с длиной элементарной единицы в 2-6 нуклеотидов).
Имеется целый набор современных технологий выявления полиморфизма на уровне ДНК, среди которых можно выделить следующие:
- анализ полиморфизма длин рестриктных фрагментов ДНК (RFLP);
- анализ полиморфизма с помощью полимеразной цепной реакции (ПЦР) и другие методы на основе амплификации ДНК между повторяющимися последовательностями в геномной ДНК.
Маркеры на основе ДНК-зондов
- RFLP (ПДРФ-маркеры, от «полиморфизм длин рестрикционных фрагментов»). Оценка полиморфизма длин рестриктных фрагментов ДНК может быть осуществлена разными способами, но наиболее традиционен метод с использованием блот-гибридизации)[1]. Этот метод включает в себя выделение ДНК, получение фрагментов рестрикции, их электрофоретическое разделение, перенос на фильтры с последующей гибридизацией специфических ДНК-зондов с полученными фрагментами ДНК. ДНК-зонд — относительно короткая последовательность клонированной ДНК с определенным уровнем гомологии и способностью гибридизоваться с соответствующим участком геномной ДНК. Комбинации рестриктаз и зондов дают высоко-воспроизводимые полиморфные спектры фрагментов ДНК, специфичные для каждого индивидуума. Различия между последними могут быть обусловлены, например, мутациями, меняющими сайт рестрикции. ПДРФ имеет ряд важных преимуществ, в числе которых высокая воспроизводимость спектров в разных лабораториях, кодоминантное «поведение» маркера. ПДРФ эффективен при картировании генома, маркировании генов многих биологических и экономически важных признаков.
- VNTR (англ. Variable Number Tandem Repeat) — метод, получивший название ДНК-фингерпринта («отпечатки пальцев»)[2]. Тандемные повторы широко распространены в разных геномах и высокополиморфны. В результате высокой вариабельности этих участков ДНК ПДРФ-анализ с зондами к микро- и минисателитным последовательностям позволяет получать мультилокусные спектры с высоким разрешением на популяционном уровне. Благодаря очень высокому уровню полиморфизма этот подход в настоящее время является хорошим инструментом для анализа внутри- и межпопуляционной изменчивости и определения генетических расстояний между группами организмов. VNTR-аллельные варианты имеют кодоминантный характер наследования.
ПЦР-маркеры
Метод полимеразной цепной реакции (ПЦР) предполагает использование специфических праймеров и получение дискретных ДНК-продуктов амплификации отдельных участков геномной ДНК. Большое количество родственных технологий построено на этом принципе. Наиболее широко используемая технология RAPD основана на анализе амплифицированных полиморфных фрагментов ДНК с помощью единичных праймеров с произвольной нуклеотидной последовательностью[3],[4],[5].
- SSR (англ. Simple Sequence Repeats) — ПЦР с флангирующими праймерами к короткому мини- или микросателлитному повтору позволяет выявлять маркеры с кодоминантным наследованием и, соответственно, удобен для выявления гетерозигот по данному локусу. Однако одна пара праймеров для флангов в ПЦР позволяет рассматривать полиморфизм только одного локуса. Для многих микросателлитных локусов не удается выявить полиморфизм. Как правило, фланкирующие последовательности для данного микросателлитного локуса оказываются видоспецифичными.
- RAPD (англ. Random Amplified Polymorphic DNA) — полимеразная цепная реакция с использованием единичного короткого (обычно 10-членного) праймера с произвольной нуклеотидной последовательностью[6],[7]. Последовательность праймеров не абсолютно любая, а ограничена значениями GC-состава 40-70 % и лингвистической сложности нуклеотидной последовательности 50-100 %. В RAPD можно использовать как одиночный праймер, так и несколько RAPD-праймеров. Продукт RAPD образуется в результате амплификации фрагмента геномной ДНК, фланкированной инвертированной последовательностью используемого праймера. Метод универсален для исследований разных видов, при использовании одних и те же праймеров. Как правило, праймер, выявляющий высокий полиморфизм для одного вида, будет эффективен и для других видов.
- ISSR (англ. Inter Simple Sequence Repeats)[8][9] — специализированный вариант метода RAPD, в котором праймер состоит из микросателлитной последовательности. В этом методе, как и в RAPD, используется один или несколько праймеров длиной в 15-24 нуклеотида. Но в данном случае праймеры состоят из тандемных коротких 2-4 нуклеотидных повторов, например, 5’-CA CA CA CA CA CA CA CA CA G, и одного или двух селективных нуклеотидов на 3’-конце праймера. Продукты ISSR-амплификации содержат на флангах инвертированную микросателлитную последовательность праймера. Так как в данном методе последовательность праймеров специфична и подбирается более строго, чем в RAPD, отжиг в ПЦР можно проводить при более высокой температуре (55-60°С), чем для метода RAPD, а поэтому фингерпринт обычно лучше воспроизводим.
- AFLP (англ. Amplified Fragment Length Polymorphism)[10] — технология, представляющая собой комбинацию методов ПДРФ и ПЦР. AFLP — сложный метод, состоящий из нескольких этапов: геномная ДНК одновременно рестрицируется двумя рестриктазами (EcoRI и MseI), узнающими 4 и 6 оснований соответственно, получая фрагменты с выступающими 3’-концами. Затем рестрицированная геномная ДНК лигируется с адаптором, содержащим «липкие» концы для рестрикционных сайтов (EcoRI и MseI). После этого проводится две последовательные ПЦР. В первой ПЦР (преамплификация) используются праймеры, полностью комплементарные адапторам EcoRI и MseI. После первой ПЦР образуется большое количество продуктов амплификации между адапторами EcoRI и MseI, которые трудно дифференцировать с помощью электрофореза. Поэтому во второй ПЦР праймеры с адапторами EcoRI и MseI содержат на 3’-конце дополнительные и не комплементарные адапторам от 1 до 3 основания, для селективной амплификации. Разделение фрагментов ДНК выполняется в полиакриламидном геле, с радиоактивной или флюоресцентной меткой соответственно.
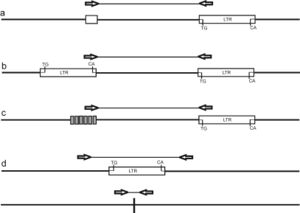
- SSAP (англ. Sequence Specific Amplification Polymorphism)[11] явился модификацией метода AFLP для выявления полиморфизма как по сайту рестрикции, так и по вставки[прояснить] в геномную ДНК транспозона или ретротранспозона. Геномная ДНК исследуемых образцов расщепляется рестриктазами PstI и MseI, и получаются фрагменты с выступающими 3’-концами. Затем рестрицированная ДНК лигируется с адапторами PstI и MseI. Первая полимеразная цепная реакция (преамплификация) проводится с праймерами от адапторов PstI и МseI, то есть амплифицируются все возможные комбинации сочетания этих адапторов в рестрицированной геномной ДНК. После первой ПЦР образуется большое количество продуктов амплификации фрагментов ДНК, локализованных между праймерами и адапторами. Продукты ПЦР разбавляются и используются для второй, селективной ПЦР. Вторая ПЦР проводится с меченным праймером к LTR и любым праймером адапторов, либо с PstI или MseI. Во второй ПЦР можно использовать праймеры к адаптору с дополнительными нуклеотидами на 3'-конце, например, один, два или три нуклеотида, не комплементарные адаптору. Электрофорез после второй ПЦР проводят в полиакриламидном геле или в секвенаторе, если использовалась флюоресцентная метка. Продукты амплификации после второй ПЦР образуются в результате амплификации фрагмента ДНК между последовательностью LTR ретротранспозона и адаптором. Получение продуктов амплификации между только последовательностями LTR принципиально возможно, но, как правило, расстояние между двумя ретротранспозонами длиннее обычно получаемых размеров продуктов ПЦР (2500 — 3000 пар оснований). А продукты амплификации между адапторами не будут выявляться, поскольку используется метка только для праймера LTR.
- IRAP Архивная копия от 30 октября 2016 на Wayback Machine (англ. Inter Retrotransposone Amplified Polymorphism)[12][13] — полимеразная цепная реакция между праймерами, комплементарными последовательностям двух рядом расположенных LTR ретротранспозона. Метод имеет несколько вариантов. В первом варианте IRAP используется единичный праймер из LTR. Продукты амплификации образуются между двумя инвертированными LTR с одинаковой последовательностью, то есть в одной цепи 5’-конец одного LTR ориентирован к 3’-концу другого LTR. Если центральная часть ретротранспозона длиннее обычного размера продуктов ПЦР (около 3000 пар оснований), то ПЦР будет проходить только между двумя LTR из разных ретротранспозиций. В этом случае соседние LTR должны располагаться в инвертированном положении. В другом варианте IRAP используются два разных праймера к инвертированным LTR: один праймер с 5’-конца, а другой с 3’-конца LTR, ориентированные в разные стороны от ретротранспозона. В данном случае соседние LTR располагаются как прямые длинные повторы. И, наконец, в третьем варианте IRAP используются праймеры к LTR из разных ретротрапозонов в различной ориентации. Можно комбинировать праймеры из LTR с другими праймерами из повторяющейся ДНК.
- REMAP (англ. Retrotransposone Microsatellite Amplified Polymorphism)[12][13] — полимеразная цепная реакция между праймером к фрагменту LTR ретротранспозона и праймером из рядом расположенного, простого микросателлитного повтора (ISSR-праймер). В данном случае позиция амплифицируемого фрагмента ретротранспозона «заякоривается» путём использования праймера к микросателлитному локусу. Например, у растений удобным оказывается использование праймера к LTR и праймера к микросателлиту (5’-CA CA CA CA CA CA CA CA CA CA G) c единичным селективным нуклеотидом на 3’-конце праймера. В REMAP применяют варианты LTR-праймеров как для 5’-конца, так и для 3’-конца LTR, как и в IRAP.
- RBIP (англ. Retrotransposon-Based Insertion Polymorphisms)[14] — метод, основанный на использовании праймеров к последовательностям ретротранспозонов и выявляющий кодоминантные аллельные варианты. Его принцип основан на мультилокусной ПЦР, в которой используются пара праймеров, фланкирующих участок ДНК до ретротранспозиции и праймер к LTR ретротранспозона, который встроен в данный участок между первыми двумя праймерами. В результате ПЦР будет амлифицироваться один из вариантов фрагментов, фланкированных парой праймеров, поскольку последовательность между LTR слишком длинная для ПЦР между сайтами геномной ДНК с ретротранспозоном внутри. Этот метод выявляет полиморфизм только для данного полиморфного локуса. К его достоинствам относят кодоминантность полиморфных вариантов, возможность использования для дот-блот-анализа большого количества сортов.
- iPBS (англ. inter PBS amplification)[15] — метод, основанный на использовании праймеров к последовательностям ретротранспозонов PBS (англ. Primer binding site, участок связывания тРНК). Метод эффективен для выявления полиморфизма между образцами, а также для клонирования новых ретротранспозонов у эукариот.
- Однонуклеотидный полиморфизм (SNP).
Примечания
- ↑ Southern E. M. Detection of specific sequences among DNA fragments separated by gel electrophoresis (англ.) // J Mol Biol[англ.] : journal. — 1974. — Vol. 98, no. 3. — P. 503—517. — doi:10.1016/S0022-2836(75)80083-0.
- ↑ Jeffreys A. J., Wilson V., Thein S. W. Hypervariable 'minisatellite' regions in human DNA (англ.) // Nature. — 1984. — Vol. 314. — P. 67—73. — doi:10.1038/314067a0.
- ↑ Kalendar R. The use of retrotransposon-based molecular markers to analyze genetic diversity (англ.) // Field and Vegetable Crops Research : journal. — 2011. — Vol. 48, no. 2. — P. 261—274. — doi:10.5937/ratpov1102261K. Архивировано 6 июля 2017 года.
- ↑ Kalendar R., Flavell A., Ellis THN, Sjakste T., Moisy C., Schulman A. H. Analysis of plant diversity with retrotransposon-based molecular markers (англ.) // Heredity : journal. — 2011. — Vol. 106. — P. 520—530. — doi:10.1038/hdy.2010.93. Архивировано 6 июля 2017 года.
- ↑ Календарь Р.Н., Глазко В.И. Типы молекулярно-генетических маркеров и их применение // Физиология и биохимия культурных растений : журнал. — 2002. — Т. 34, № 4. — С. 141—156. (недоступная ссылка)
- ↑ Williams J. G., Kubelik A. R., Livak K. J., Rafalski J. A., Tingey S. V. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers (англ.) // Nucleic Acids Research[англ.] : journal. — 1990. — Vol. 18, no. 22. — P. 6531—6535. — doi:10.1093/nar/18.22.6531.
- ↑ Welsh J., McClelland M. Fingerprinting genomes using PCR with arbitrary primers (англ.) // Nucleic Acids Research[англ.] : journal. — 1990. — Vol. 18. — P. 7213—7218. — doi:10.1093/nar/18.24.7213.
- ↑ Сиволап Ю.М., Календарь Р.Н., Чеботарь С.В. Генетический полиморфизм злаковых растений при помощи ПЦР с произвольными праймерами // Цитология и Генетика : журнал. — 1994. — Т. 28. — С. 54—61. Архивировано 4 марта 2016 года.
- ↑ Zietkiewicz E., Rafalski A., Labuda D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification (англ.) // Genomics : journal. — Academic Press, 1994. — Vol. 20, no. 2. — P. 176—183. — doi:10.1006/geno.1994.1151.
- ↑ Vos P., Hogers R., Bleeker M., Reijans M., van de Lee T., Hornes M., Frijters A., Pot J., Peleman J., Kuiper M., et al. AFLP: a new technique for DNA fingerprinting (англ.) // Nucleic Acids Research[англ.] : journal. — 1995. — Vol. 23. — P. 4407—4414. — doi:10.1093/nar/23.21.4407.
- ↑ Waugh R., McLean K., Flavell A. J., Pearce S. R., Kumar A., Thomas B. B., Powell W. Genetic distribution of Bare-1-like retrotransposable elements in the barley genome revealed by sequence-specific amplification polymorphisms (S-SAP) (англ.) // Molecular General Genetics : journal. — 1997. — Vol. 253, no. 6. — P. 687—694. — doi:10.1007/s004380050372.
- ↑ 12,0 12,1 Kalendar R., Grob T., Regina M., Suoniemi A., Schulman A. H. IRAP and REMAP: Two new retrotransposon-based DNA fingerprinting techniques (англ.) // Theoretical and Applied Genetics[англ.] : journal. — 1999. — Vol. 98. — P. 704—711. — doi:10.1007/s001220051124. Архивировано 4 марта 2016 года.
- ↑ 13,0 13,1 Kalendar R., Schulman A. H. IRAP and REMAP for retrotransposon-based genotyping and fingerprinting (англ.) // Nature Protocols[англ.] : journal. — 2006. — Vol. 1, no. 5. — P. 2478—2484. — doi:10.1038/nprot.2006.377. Архивировано 9 ноября 2016 года.
- ↑ Flavell A. J., Knox M. R., Pearce S. R., Ellis THN. Retrotransposon-based insertion polymorphisms (RBIP) for high throughput marker analysis (англ.) // The Plant Journal[англ.] : journal. — 1998. — Vol. 16, no. 5. — P. 643—650. — doi:10.1046/j.1365-313x.1998.00334.x.
- ↑ Kalendar R., Antonius K., Smykal P., Schulman A. H. iPBS: A universal method for DNA fingerprinting and retrotransposon isolation (англ.) // Theoretical and Applied Genetics[англ.] : journal. — 2010. — Vol. 121, no. 8. — P. 1419—1430. — doi:10.1007/s00122-010-1398-2. Архивировано 9 ноября 2016 года.